서 론
시각화-데이터 요약
연속형 변수 요약 - 상자그림, 히스토그램

범주형 변수 요약 - 막대그래프, 원그래프

시각화-데이터 탐색
주성분 분석

히트맵

네트워크 시각화
생존자료의 시각화
Figure 4.


시각화-예측모형 평가 및 해석
예측모형평가 - ROC Curve
Figure 5.

AbstractWith the advancement and acceleration of digital technology, the demand and supply of healthcare big data are increasing. In Korea, the government and companies have made various efforts to utilize healthcare big data, such as deregulation data-related legal regulations and data linkage between different institutions. As a result, many researchers have been able to access a variety of healthcare big data. Although healthcare big data has a vast amount and high value, many researchers are unable to fully access healthcare big data because there are difficulties in processing, analysis, and interpretation for data. The data visualization is recognized as an important tool that can solve these limitations. Using data visualization, researchers can intuitively understand complex data and receive support for decision-making. Additionally, these visualizations promote effective communication between experts in different fields and between experts and non-experts. Visualization is used in a variety of research fields and processes, including data summarization, data exploration, and evaluation and interpretation of predictive models. Various types of visualization have different meanings depending on how they are expressed. Therefore, it is important to express the meaning of visualization appropriately. This study provides representative examples of visual representations for data summarization, data exploration, and predictive model evaluation. This study aimed to improve easier access and utilization of healthcare big data by providing R code and visualization results.
서 론최근 디지털 기술의 발전과 혁신으로 데이터의 양이 크게 증가하고 고도화되고 있으며, 보건의료 분야에서도 스마트폰, 웨어러블 기기, Internet of Things (IoT), 소셜미디어 등 여러 가지 데이터 소스를 통해 실시간 단위의 건강정보, 활동기록과 같은 헬스케어 정보가 데이터화되어 축적되고 있다[1,2]. 정부와 기업은 효율적인 보건의료빅데이터의 활용을 위해 전문 인력 양성, 기술 개발 및 인프라 구축에 큰 투자를 하고 있다. 가명정보 결합 제도의 완화, 의료데이터 개방 및 활용 촉진 등 국내 데이터 개인정보 보호 및 법적 규제들이 완화되었다[3-5]. 그 결과, 많은 연구자들에게 다양한 기관들의 데이터가 공개되었고 기관 간의 연계가 가능해지면서, 보건의료 분야의 혁신 및 발전, 글로벌 경쟁력 강화, 효율적인 서비스 제공 등 보건의료빅데이터는 더욱 큰 잠재력을 가지게 되었다[6,7].
하지만 보건의료빅데이터는 방대한 양과 높은 활용가치를 지니고 있음에도 불구하고 많은 연구자들이 이를 완전히 활용하지 못하고 있다[3]. 대부분의 데이터가 현재 의료기관이나 기업에 의해 소유된 형태이기 때문에 라이선스가 필요하거나 비용이 요구되며[8,9], 보건의료빅데이터가 활용되지 못하는 가장 큰 이유 중 하나는 여러 전문 지식이 필요하다는 점이다. 생물학적 관계나 보건의료 분야에 대한 전문 지식이 부족한 경우, 다양한 요인들이 복잡한 관계를 가지고 있어 정확한 해석을 기대하기 어렵다. 또한, 보건의료 분야는 개인이나 집단의 건강과 직결되기 때문에 잘못된 해석에 대한 위험성이 높다[10]. 그리고 보건의료빅데이터는 다양한 형태와 소스에서 생성되고, 이에 따른 데이터 크기와 복잡성이 존재하기 때문에 데이터 수집, 처리 및 분석에 필요한 통계 관련 기술이 요구된다[11-13].
데이터 시각화는 여러 장점을 가지고 있어, 복잡하고 다양한 형식의 데이터를 이해하고 유용한 정보를 추출하는 중요한 도구로 인식되고 있다. 먼저, 데이터 시각화는 빅데이터를 직관적으로 이해할 수 있고, 패턴, 경향 및 이상치를 신속하게 발견하는 데에 이점이 있다[14]. 두 번째, 빅데이터의 시각화는 의사 결정을 지원하는 데에 중요한 역할을 하며, 데이터 간의 관계와 패턴을 발견함을 통해 이를 기반으로 전략을 개발하거나 문제를 해결할 수 있게 된다[3,15]. 마지막으로, 시각화는 정보의 대중화, 객관적 전달, 정보에 대한 접근성 강화 등에서 매우 효율적이고 직관적인 행위이기 때문에 보건의료 및 생물학적 지식이 없더라도 데이터 간의 일부 관계를 파악할 수 있다[16]. 또한, 시각화된 결과는 데이터 속에서 정보를 찾아 직관적으로 정보를 전달하는 측면에서 비전문가뿐만 아니라 전문가와의 효과적인 커뮤니케이션을 촉진할 수 있다[17].
본 논문에서는 다양한 연구자들이 보건의료빅데이터를 더 쉽게 접근하고 분석에 활용할 수 있도록 다양한 시각화 방법을 정리하고 설명하였다. 시각화 방법들을 설명하기 위해 R version 4.3.2 (R foundation for Statistical Computing, Vienna, Austria) 프로그램을 사용하였고, 데이터는 R의 ‘ survival’ 패키지에서 제공하는 ‘ colon’ 데이터와 ‘ heplots’ 패키지에서 제공하는 ‘ Diabetes’ 데이터를 사용하여 분석하였다. 분석에 사용된 시각화 방법은 직접 구현할 수 있도록 R 코드를 제공하고자 한다.
시각화-데이터 요약시각화를 통해 데이터의 요약된 정보를 추정하고 파악할 수 있다. 데이터 분석 초기 단계에서 데이터의 풍부한 정보를 빠르게 이해하는 데에 중요한 역할을 하며, 비율, 평균, 중앙값, 표준편차, 분위수와 같은 중요한 통계 지표를 추정하고 비교할 수 있다. 이러한 통계량은 데이터의 특성을 요약하고 특성 간의 관계를 파악할 수 있을 뿐만 아니라 분포 가정을 통해 분석 방법을 결정하는 역할이 가능하다. 본 절에서는 데이터 요약을 크게 연속형 변수와 범주형 변수로 나누어 기본적인 시각화 방법을 소개하고 ‘ ggplot2’ 패키지를 활용한 R 코드와 시각화 그림을 제공한다.
연속형 변수 요약 - 상자그림, 히스토그램탐색적 데이터 분석에서 주로 사용하는 방법 중에 하나는 ‘상자그림(box plot)’으로 연속형 변수를 대상으로 사용한다. 일반적인 구성은 Figure 1A처럼 중앙값, 사분위수, 최저 및 최고점을 볼 수 있으며, 데이터 분포의 수준, 산포 및 대칭성에 대한 정보를 전달한다. 이 5가지의 값의 모음은 데이터 세트의 분포를 요약하는 빠른 방법이며 이 특성에 대한 값을 통해 데이터를 비교하는 간단한 방법을 제공한다. 상자그림의 상자는 사분위수의 상위와 하위의 위치를 나타내는 데 사용되며, 이는 분포의 50%를 구성한다. 또한, (사분위수 범위*1.5) 외의 범위 밖의 값인 이상치의 정보를 제공하여 이 값들을 가진 대상자의 특성을 확인하거나 제거할 수 있다[18,19]. 히스토그램(histogram)은 연속적인 데이터의 분포를 시각적으로 나타내는 데 사용되는 그래프 형식 중 하나로, 데이터의 빈도 분포를 막대 모양의 기둥으로 표현하여 데이터의 분포, 중심 경향, 분산 등을 이해하고 탐색하는 데 유용하다. 또한, 히스토그램에서는 커널 밀도(Kernel density estimation)와 같은 부드러운 곡선 표현을 통해 데이터의 분포 추정이 가능하기 때문에, 가우시안 분포, 포아송 분포 등 분포 가정에 따른 적합한 통계 모형 선택이 가능하다(Figure 1B) [20].
 범주형 변수 요약 - 막대그래프, 원그래프막대그래프(bar plot)와 원그래프(piechart)는 범주형 데이터의 빈도나 크기를 나타내는 시각화 도구이다. 막대그래프는 비교, 분포, 패턴, 추이 등을 쉽게 파악할 수 있도록 도와주고, 해석이 비교적 간단하고 직관적이기 때문에 데이터 분석과 보고서 작성에 유리하다. 원그래프는 전체 집합에서 각 부분의 상대적인 크기를 나타내기 위한 그래프로, 원 모양 내에 범주형 변수의 범주별 비율을 시각적으로 확인할 수 있다. Figure 1C와 Figure 1D는 각각 막대그래프와 원그래프로, ‘ survival’ 패키지에서 제공하는 ‘ colon’ 데이터의 나이에 따른 상태와 성별 빈도를 시각화하여 표현하였다.
 시각화-데이터 탐색보건의료빅데이터에서 데이터에 대한 사전 정보가 없거나 제한적인 경우에 데이터 시각화는 데이터의 핵심 특성과 상호 관계를 직관적으로 탐색할 수 있는 강력한 도구이다. 또한, 시각화된 자료로부터 숨겨진 규칙과 패턴을 발견하여 의미가 있는 정보를 습득하거나 새로운 해석이 가능하게 할 수 있다. 데이터 탐색을 위한 시각화 기법은 빅데이터 분석의 가설 설정, 연구 설계, 자료수집, 자료분석 등 여러 측면에서 데이터 기반의 의사 결정을 지원한다. 그리고 한눈에 데이터 내 객체 및 집단의 특성이나 변수들의 상호 관계를 볼 수 있어 데이터 간 비교나 데이터 변화를 추적하는 데도 유용하다.
주성분 분석주성분 분석(principle component analysis, PCA)은 서로 상관되어 있는 변수들 간의 복잡한 구조를 분석하기 위하여 p개의 변수들을 선형변환시켜 주성분이라고 불리는 서로 상관되어 있지 않은(혹은 독립적인) 새로운 인공변수들을 생성하는 기법으로, 요약하자면 고차원 변수들을 저차원 데이터로 환원시키는 기법이다. 여러 변수들 x=(x1,x2,…,ap)' 에 대해 (1)과 같은 형식의 가중결합을 취한 것을 선형변환이라고 하는데 이때 변환값 y는 가중계수(weight)들인 벡터 a = (a1,a2,…,xp)' 의 적절한 선택에 따라 원래의 x보다 더 유용한 정보를 보유할 수 있고, 차원의 축소를 통해 관계를 단순화시키는 데에 큰 역할을 한다. 즉, 주성분 분석은 많은 변수들의 정보를 2차원, 3차원 상으로 표현할 수 있기 때문에 데이터 시각화에 매우 유용하다[21].
Figure 2의 (A)와 (B)는 각각 R의 ‘ ggplot2’ 패키지를 사용해서 주성분 분석의 Scree plot과 객체들을 2차원으로 표현한 산점도 그림이다. 먼저 Scree plot는 주성분의 개수를 결정하는 데 중요한 시각화 방법 중 하나로 주성분의 설명력과 주성분 개수 사이의 관계를 시각적으로 나타낸다. 시각화 결과를 요약하면 주성분 분석에 포함되는 변수들을 설명하는 정도가 주성분 1이 86.6%, 주성분 2가 10.9%으로, 두 주성분을 통해 5가지의 변수의 약 97.5%가 설명된다고 할 수 있다. 적절한 주성분 개수는 보통 누적비율의 합이 80%나 90%가 초과하는 개수를 기준으로 한다[22]. 산점도 그림은 선택된 주성분 두 개를 2차원 좌표로 하여 객체들을 시각화한 그래프로, 당뇨 진단 여부(정상, 화학적 당뇨, 명백한 당뇨)에 따라 색을 달리하여 표시한 결과이다. 직관적으로 집단 간 개체 사이에 유사한 패턴을 가지고 있음을 확인할 수 있고, 더 깊은 해석도 가능하다. 시각화 그림에서 당뇨 진단 여부는 PC1 (주성분 1) 크기에 따라 분류할 수 있기 때문에, PC1은 당뇨 진단 여부를 분류하고 예측에 중요한 변수로 활용이 가능하다. 추가적으로 주성분의 가중계수(eigenvalue) 확인을 통해 가중치 높은 변수들 각각을 당뇨의 위험인자로써의 평가가 가능하다(Supplementary Table 1). PCA를 통한 시각화는 임상적인 지식이 요구되지 않고, 데이터를 통해 위험요인을 평가하고 인과성 추론이 가능하기 때문에 많은 연구자들이 활용하고 있다[23-25].
 히트맵행과 열로 구성된 데이터 세트의 관계를 시각화하는 데 유용한 시각화 도구로 히트맵(heatmap)이 있다[26]. 히트맵은 열을 뜻하는 히트와 지도를 뜻하는 맵을 결합시킨 단어로, 데이터의 상대적인 값을 색상으로 나타내어 패턴, 추세, 높낮이 등을 파악하기에 유용하다. 히트맵은 덴드로그램(dendrogram)과 함께 주로 사용되는데, 덴드로그램은 요소와 요소, 객체와 객체의 관계를 거리(distance)를 바탕으로 Hi-erarchical cluster를 덴드로그램 형태로 표현함으로써 개체 및 성분 간 군집화 양상도 함께 보여줄 수 있어 유전체 빅데이터 분석에서도 시각화 방법으로 자주 활용한다.
Figure 2의 (C)는 R 프로그램의 ‘ pheatmap’ 패키지를 활용하여 Diabetes 데이터를 히트맵을 통해 탐색한 결과이다[27]. 그 결과, 정상군에서는 다른 군들에 비해 sspg (인슐린 저항도), relwt (상대 체중)가 상대적으로 낮았으며, 화학적 당뇨군에서는 relwt (상대 체중), instest (경구 포도당에 대한 인슐린 반응), 명백한 당뇨군에서는 sspg, glufast (공복 혈당), glutest (포도당 불내성)가 상대적으로 높은 패턴을 보였다. 또한, 덴드로그램을 통해 요소와 요소의 평균 간의 연관성을 확인한 결과 glufast와 glutest, relwt와 instest 사이에서 높은 연관성을 보인다.
 네트워크 시각화네트워크 시각화(network visualization)는 데이터나 시스템의 연결을 그래픽으로 나타내는 프로세스이다. 네트워크 시각화의 기초는 그래프 이론인데, 노드와 노드 사이의 연결로 구성되어 노드 간의 관계를 나타낸다. 네트워크 시각화는 보건의료빅데이터 분석에서 다양한 의료 정보 및 데이터 요소 간의 상호작용, 관계, 패턴을 시각적으로 나타내기 위해 주로 활용된다[28]. 또한, 원데이터를 세분화한 각 데이터에서 변수들 간의 관계가 어떻게 달라지는지 시각적인 비교가 가능하다.
Figure 3은 Diabetes 데이터를 group의 범주에 따라 데이터를 세분화하여 각 데이터에서 변수 간의 상관분석을 통해 상관관계를 확인하고, 이를 시각화한 그림이다. 각 데이터별로 변수 간의 상호관계가 다르다는 것을 시각적으로 볼 수 있다. 정상군의 네트워크 시각화에 대한 결과를 보면 glutest (포도당 불내성)은 relwt (상대 체중)과 sspg (인슐린 저항성)과 높은 상관관계를 가지기 때문에 정상군에게서는 인슐린 저항성과 상대 체중을 관리하면 포도당 불내성에 대한 관리가 가능하다는 의사결정을 지원한다. 또한, 화학적 당뇨 집단에서 비슷한 시각적인 결과가 도출되었지만, 정상군에서의 네트워크 시각화 결과와는 다르게 상관성이 약해진 것을 볼 수가 있다. 그리고 명백한 당뇨군에서는 모든 변수들 간 상관관계가 일정한 것을 볼 수가 있는데, 이는 이미 수치가 높아질 만큼 높아져서 관계가 없어 보이거나, 변수 간에 독립성이 존재한다고 데이터를 미리 이해하고 접근할 수 있다. Figure 3에 대한 R 코드는 Supplementary Contents 1에 첨부하였다.
생존자료의 시각화생존분석은 환자의 관심 사건이 발생하기까지 걸린 시간을 분석하는 통계적 방법론으로 사건 발생 확률과 생존 확률을 추정한다. 보건의료빅데이터는 전자 건강 기록(electronic health records, EHRs), 의료 청구 데이터, 생물학적 데이터, 환자 의견 및 만족도 등 생존 분석에 필요한 대량의 정보를 제공하며, 환자 그룹 간 생존 패턴 및 예측 변수의 효과를 평가하는 데 사용되어 생존분석과 보건의료빅데이터는 불가분의 관계이다. 이러한 생존분석은 특정 질병이나 조건과 관련된 위험 요인을 식별하고, 예방 또는 조치를 취하기 위한 정보를 제공한다. 또한 최근 딥러닝의 발전으로 DeepHit, DeepConvSurv, WSISA 등 딥러닝 신경망 모형을 통한 생존분석을 이용하여 기존 생존 모형에서의 한계점인 복잡한 시간 종속성과 사건 간의 관계가 보완되었다. Figure 4는 여러가지 형태의 모든 생존 모형에서 데이터 탐색을 위한 대표적인 시각화 방법인 카플란-마이어(Kaplan-Meier) 플롯과 누적발생함수(cumulative incidence function, CIF) 플롯으로, 전체 대상자의 추적 기간과 건강 상태를 평가하고 질병 발생률 모니터링이 가능하다. 생존분석을 위한 시각화를 위해 R의 ‘ survival’ 패키지를 통해 분석을 수행하고, 패키지 내 제공하는 ‘ lung’ 데이터를 사용하여 폐암 환자에서 시간에 따른 생존율을 성별끼리 비교할 수 있도록 시각화하였다.
Figure 4.(A) Kaplan-Meier survival plot and (B) Cumulative incidence function in survival analysis. 
 시각화-예측모형 평가 및 해석예측모형 개발은 보건의료 분야에서 질병의 진단, 치료를 위해 활발히 연구되고 있는 분야이다. 또한, 2010년 이후부터 전자의무기록(electronic medical record, EMR)의 보급률이 높아지고 데이터 관리 기술이 발달함에 따라 대부분의 상급종합병원과 종합병원은 긴 기간 동안의 환자 처방 및 검사 결과 정보를 전산화하고 저장하고 있기 때문에 단일 병원 자료로도 머신러닝, 딥러닝 알고리즘을 이용한 연구가 활발하다[29,30].
추가적으로 정부는 마이헬스웨이(의료 마이데이터), 네트워크 사업 등 여러 사업들을 통해 다양한 건강 정보를 수집할 수 있도록 하였고, EHR과 personal healthcare record (PHR) 같은 환자의 의료 기록과 치료 정보가 추적이 가능해짐에 따라 데이터의 양은 더욱더 증가하게 되었다[31]. 이러한 데이터의 증가와 고도화는 예측모형 연구의 양과 질 향상에 깊은 연관성을 가지고 있고, 기존 통계모형에서 복잡한 머신러닝, 딥러닝의 구조를 사용할 때 발생할 수 있는 문제들을 극복할 수 있다. 방대한 훈련 데이터의 양은 예측모형의 주요 문제점인 과적합 현상을 방지하고, 더 일반적인 패턴을 학습할 가능성이 증가하여 높은 타당성과 신뢰성을 가진다.
하지만, 데이터의 양이 증가함에 따라 예측모형에 대한 활용도가 높아졌음에도 아직까지 많은 연구자들에게 머신러닝, 딥러닝 예측모형에 대한 진입장벽은 높다. 또한, 모형의 복잡한 구조로 인해 발생하는 ‘블랙박스(black-box)’ 현상 때문에 예측모형에 대한 해석의 어려움을 겪고 있다[32]. 본 절에서는 시각적인 방법을 통해 모델이 높은 설명력을 가지는지 평가하는 방법과 블랙박스 현상을 해결할 수 있는 설명가능한 AI (eXplainable AI) 중 하나인 SHAP plot을 제시하였다[33].
예측모형평가 - ROC Curve의학 분야에서 주로 질병 진단이나 의료 영상 분석에서 예측 또는 분류 모델의 성능을 평가할 때, receiver operation characteristic curve (ROC) 곡선은 시각적으로 모델의 성능을 평가하고 여러 모델의 설명력을 비교하기 위해 많이 사용된다. ROC 곡선은 true positive rate (TPR)의 변화에 따른 false positive rate (FPR)의 변화를 직관적인 곡선 형태로 보여준다. 여기서 TPR은 실제 양성을 양성으로 예측하는 비율인 민감도를 의미하고, FPR은 실제 음성을 양성으로 잘못 예측하는 비율인 특이도를 의미한다. ROC 곡선은 좌측 상단에 붙어 있을수록 모형의 성능이 좋은 것으로 판단할 수 있고, ROC 곡선 아래 영역을 나타내는 area under the curve (AUC)를 통해 분류 모델의 전체적인 성능을 평가할 수 있다. AUC를 구하는 식은 다음과 같이 나타낼 수 있다[34].
이 AUC 크기가 작을 수록 모델의 성능이 낮을 것을 의미하며, 1에 가까울수록 완벽한 모델이라고 해석한다(Supplementary Table 2).
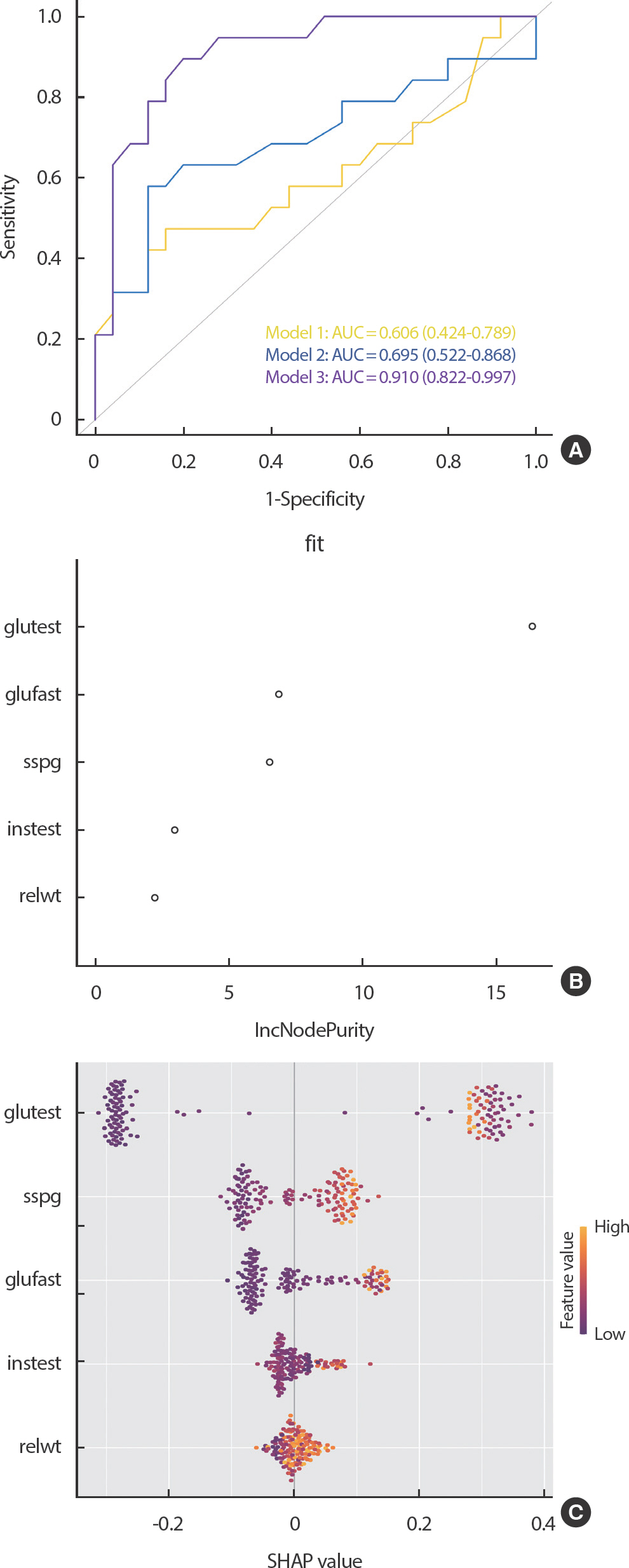
Figure 5의 (A)는 Diabetes 데이터에서 머신러닝 모형 중 하나인 랜덤포레스트 모형을 활용하여 당뇨를 예측하기 위해 구축한 모델 Model 1, Model 2, Model 3에 대한 ROC 곡선이다. 먼저, 당뇨를 정의하기 위해 group 변수가 Normal인 개체를 0 (Normal), Chemical_Diabetic 또는 Overt_Diabetic인 개체를 1 (diabetes)로 나누어 새로운 변수 diabe-tes를 생성하였고, 예측모형을 평가하기 위해 데이터를 70:30의 비율로 훈련 데이터와 테스트 데이터로 나누어 학습과 평가하였다. ROC 곡선의 시각화는 R의 ‘ pROC’ 패키지를 사용하였다.
Figure 5.ROC curve, variable importance plot, and SHAP plot for prediction model from Diabetes data. ROC, receiver operation characteristic; SHAP, SHapley Additive exPlanations. 
Model 1은 하나의 예측 변수(instest)만을 가지고 당뇨군을 예측한 모델이며, Model 1에 relwt, sspg를 (순차적으로) 추가한 Model 2와 Model 3를 구축하였다. ROC 곡선을 통해 Model 1에서 Model 3으로 갈수록 곡선이 좌측 상단에 가까워지는 것을 확인할 수 있고, 이는 모델에 변수가 하나씩 추가될수록 모델의 예측력(설명력)이 증가하는 것을 시각적으로 보여준다. 또한, 각 ROC 곡선 사이의 간격을 보았을 때, Model 2와 Model 3 사이의 곡선 간격이 Model 1과 Model 2의 사이의 간격보다 더 넓은 것을 확인할 수 있다. 이는 Model 3에 추가된 sspg 변수가 당뇨군을 예측하는데 더 강력한 예측력을 가진 변수로써 추측할 수 있다.
 eXplanable AI (xAI) - SHAP plot기존 예측모형을 평가하기 위한 변수중요도 플롯(variable importance plot)은 구축된 예측모형에 포함된 특성들의 중요도를 평가하고 특성 선택 및 모델 향상에 도움을 주는 데 사용되었다. 그러나 변수중요도 플롯은 블랙박스 모델(예, 신경망, 랜덤포레스트, 그래디언트 부스팅 등)에 대한 중요도는 보여줄 수 있지만, 어떤 특성이 어떻게 결정에 기여하는지 정확히 설명하기 어려운 단점이 있다(Figure 5B) [35].
SHapley Additive exPlanations (SHAP) plot은 모델의 예측을 해석하고, 각 특성이 예측에 어떻게 기여하는지 시각적으로 표현하기 위한 가장 대중적인 xAI 방법 중 하나이다. SHAP plot은 게임 이론의 Shapley value의 개념을 머신러닝 예측 모델의 해석에 적용하여 시각화한 그래프로, Shapley value의 수식은 (3)으로 표현된다.φi는i 번째 데이터에 대한 Shapley value, F는 전체 집합, S는 모델에 사용된 특성들의 부분집합,fs∪i(χs∪i) 는 번째 데이터를 포함한 전체 기여도를 나타내고,fs(χs) 는 번째 데이터를 제외한 나머지 부분 집합의 기여도를 나타낸다.
이 Shapley value는 가능한 모든 특성의 조합에 대해 모델의 예측 결과를 계산하고 이를 평균화함으로써, 각 특성이 개별 예측에 어떻게 영향을 미치는지 보여주는데, 이것을 시각화 방법으로써 표현한 것이 SHAP Plot이다. 이러한 SHAP Plot은 Summary plot, Dependency plot, Force plot 등 여러 시각화 방법을 통해 다양한 해석을 제공하기 때문에 연구자의 의도에 따라 각 사용 용도에 따라 활용이 가능하다는 장점이 있다.
Figure 5의 (C)는 SHAP plot의 Summary plot 중 kernelSHAP를 사용하여 시각화한 그림이다. Summary plot 중에는 TreeSHAP가 대표적이지만 랜덤포레스트 모형은 많고 깊은 tree가 존재하여, 단순히 Tree-SHAP로 처리하게 되면 엄청난 양의 메모리와 CPU 성능이 필요하기 때문에 모든 예측모형에 작동하는 KernelSHAP를 사용하였다.
고 찰본 연구는 보건의료빅데이터 연구에서 시각화하는 방법에 대한 주요 사례들을 제시하였다. 보건의료 분야에서 점차 방대해지는 데이터를 어떻게 처리하고, 해석할지는 여전히 하나의 과제로 남아 있다.
보건의료 분야에서 부족한 지식을 제시된 시각화 기법들을 통해 데이터 요약, 탐색, 예측 모형 평가로 나눠 다양한 정보들과 통찰력을 얻을 수 있었다. 시각화를 통한 해석은 해석자에 대한 주관성, 사용자의 경험과 지식, 의도와 목적에 따라 다르게 나타난다. 따라서, 사용자의 목적에 따라 데이터를 다양한 관점에서 고려하면 의사, 정책 제안자, 연구자 등은 자신의 목표에 맞는 정보를 더 정확하게 얻을 수 있으며 이를 기반으로 개인화된 의사결정을 내릴 수 있다. 또한, 시각화를 통해 얻어진 정보는 타인에게 공유가 가능하고 쉽게 설명 가능하다는 장점이 있고, 예상치 못한 패턴, 관계, 혹은 문제점에 대한 추가적인 정보를 습득할 수 있다. 이는 새로운 가설과 우선순위 설정, 효과적인 전략 수립 등 여러 방법으로 활용될 것이다.
Supplementary MaterialSupplementary Table 1.The eigenvectors for principle component analysis
REFERENCES1. Schroeck M, Shockley R, Smart J, Romero-Morales D, Tufano P. Ana-lytics: The real-world use of big data. IBM Global Business Services 2012;12:1-20.
2. Ok JA, Cho MS. A study on the role & governance of Gyeonggi-do bigdata center. Suwon: Gyeonggi Research Institute; 2015. p. 1-153. (Korean).
3. Bak SH, Lee HM, Lee JB, Bae JH. Reinterpreting and utilizing data visualization based on public data. Proceedings in the Korea Information Processing Society Conference. 2017;791-794.
4. Lee Y. A study on the revision trend of data 3 act. J Comp Priv Law 2020;27(2):423-463. (Korean).DOI: 10.22922/jcpl.27.2.202005.423.
5. Kang TW. Data 3 Act passed… Prospects for resilience in industries such as medical and AI. KISO J 2020;38:25-29. (Korean).
6. Lee JH, Je MK, Cho MJ, Son HS. Trends in using big data in the healthcare field. J Korean Inst Commun Inf Sci 2014;32(1):63-75. (Korean).
7. Lim GC. Health and welfare data utilization status and challenges. Health Welf Policy Forum 2020;2020(8):2-5. (Korean).
8. National Health Insurance Service. NHISS (National Health Insurance Sharing service). Available at https://nhiss.nhis.or.kr/bd/ay/bdaya001iv.do [accessed on September 9, 2023].
9. Health Insurance Review and Assessment Service. HIRA Bigdata Open portal. Available at https://opendata.hira.or.kr/home.do [accessed on September 9, 2023].
10. Noh YS. Expression of HIRA big data: Toward intelligent visualization analysis and two-way communication. HIRA Issue 2019;11:1-6. (Korean).
11. Rumsfeld JS, Joynt KE, Maddox TM. Big data analytics to improve cardiovascular care: Promise and challenges. Nat Rev Cardiol 2016;13(6):350-359. DOI: 10.1038/nrcardio.2016.42.
12. Slobogean GP, Giannoudis PV, Frihagen F, Forte ML, Morshed S, Bhan-dari M. Bigger data, bigger problems. J Orthop Trauma 2015;29(Suppl 12):S43-46. DOI: 10.1097/BOT.0000000000000463.
13. Chang EJ, Kim DH, Lee JY, Yang BR, Hwang JS, Kwak SK, et al. Research on ways to advance the use of healthcare big data. Wonju: Health Insurance Review and Assessment Service; 2016. p. 1-81. (Korean).
14. Kim H. Case study of bigdata visualization-centre around the visual representation form-. J Integr Design Res 2014;13(4):125-136. (Korean).DOI: 10.21195/jidr.2014.13.4.011.
15. Oh JH, Kim DJ, Kim JD. Big data visualization methods and visualization process. J Korea Multimed Soc 2014;18(1):24-31. (Korean).
16. Ahn CM. A study on the current status of data visualization. Proceedings of the KICS Summer Conference. 2016;764-765.
17. Yoon JH, Cho JK. A study on information visualization using data science. J Cult Prod Design 2020;61:11-20. (Korean).
18. Williamson DF, Parker RA, Kendrick JS. The box plot: A simple visual method to interpret data. Ann Intern Med 1989;110(11):916-921. DOI: 10.7326/0003-4819-110-11-916.
19. Potter K, Hagen H, Kerren A, Dannenmann P. Methods for presenting statistical information: The box plot. Visualization of Large and Unstructured Data Sets (VLUDS). 2006;97-106.
20. Ding N, Zhou SD, Sun ZQ. Histogram-based estimation of distribution algorithm: A competent method for continuous optimization. J Com-put Sci Technol 2008;23(1):35-43. DOI: 10.1007/s11390-008-9108-0.
21. Bro R, Smilde AK. Principal component analysis. Anal Methods 2014;6(9):2812-2831. DOI: 10.1039/c3ay41907j.
22. Jolliffe IT. Principal component analysis for special types of data. Principal Component Analysis. Springer Series in Statistics. New York: Springer; 2002. p. 338-372.
23. Khamis GSM, Alanazi SM. Exploring sex disparities in cardiovascular disease risk factors using principal component analysis and latent class analysis techniques. BMC Med Inform Decis Mak 2023;23(1):101. DOI: 10.1186/s12911-023-02179-3.
24. Zhao J, Guo W, Wang J, Wang T. Exploring the association of dietary patterns with the risk of hypertension using principal balances analysis and principal component analysis. Public Health Nutr 2023;26(1):160-170. DOI: 10.1017/S136898002200091X.
25. Kherif F, Latypova A. Principal component analysis. Machine Learning. Cambridge: Academic Press; 2020. p. 209-225.
26. Wilkinson L, Friendly M. The history of the cluster heat map. Am Stat 2009;63(2):179-184. DOI: 10.1198/tas.2009.0033.
27. Kolde R. Package ‘pheatmap’. Available at https://cran.r-project.org/web/packages/pheatmap/pheatmap.pdf [accessed on September 8, 2023].
28. Iandolo F, Vito P, Fulco I, Loia F. From health technology assessment to health technology sustainability. Sustainability 2018;10(12):4748. DOI: 10.3390/su10124748.
29. Yoon D, Chang BC, Kang SW, Bae H, Park RW. Adoption of electronic health records in Korean tertiary teaching and general hospitals. Int J Med Inform 2012;81(3):196-203. DOI: 10.1016/j.ijmedinf.2011.12.002.
30. Kim YG, Jung K, Park YT, Shin D, Cho SY, Yoon D, et al. Rate of electronic health record adoption in South Korea: A nation-wide survey. Int J Med Inform 2017;101:100-107. DOI: 10.1016/j.ijmedinf.2017.02.009.
31. Jung IY, Choi BS, Song MJ, Kim JE. A study on the integrated strategy for utilizing healthcare data public platform. Sejong: Science and Tech-nology Policy Institute; 2021. p. 1-273. (Korean).
32. Ching T, Himmelstein DS, Beaulieu-Jones BK, Kalinin AA, Do BT, Way GP, et al. Opportunities and obstacles for deep learning in biology and medicine. J R Soc Interface 2018;15(141):20170387. DOI: 10.1098/rsif.2017.0387.
33. Ribeiro MT, Singh S, Guestrin C. “Why should i trust you?” Explaining the predictions of any classifier. KDD ‘16: The 22nd ACM SIG-KDD International Conference on Knowledge Discovery and Data Mining. 2016 Aug 13-17;San Francisco.1135-1144.
|
|
|||||||||||||||||||||||||||||||||||||