서 론
연구 방법
연구 개요
연구대상 및 자료수집
Table 1.
Table 2.
항암제 분류
Table 3.
항암제 세대-암 병기별 생존 시간 히스토그램
변수에 따른 생존율 산포도
카플란-마이어(Kaplan-Meier) 생존분석
콕스(Cox) 비례위험 모형
성향 점수 매칭
연구 결과
분석데이터
사망자분포: 항암제 세대 및 암 병기별 분석
생존분석
Figure 4.
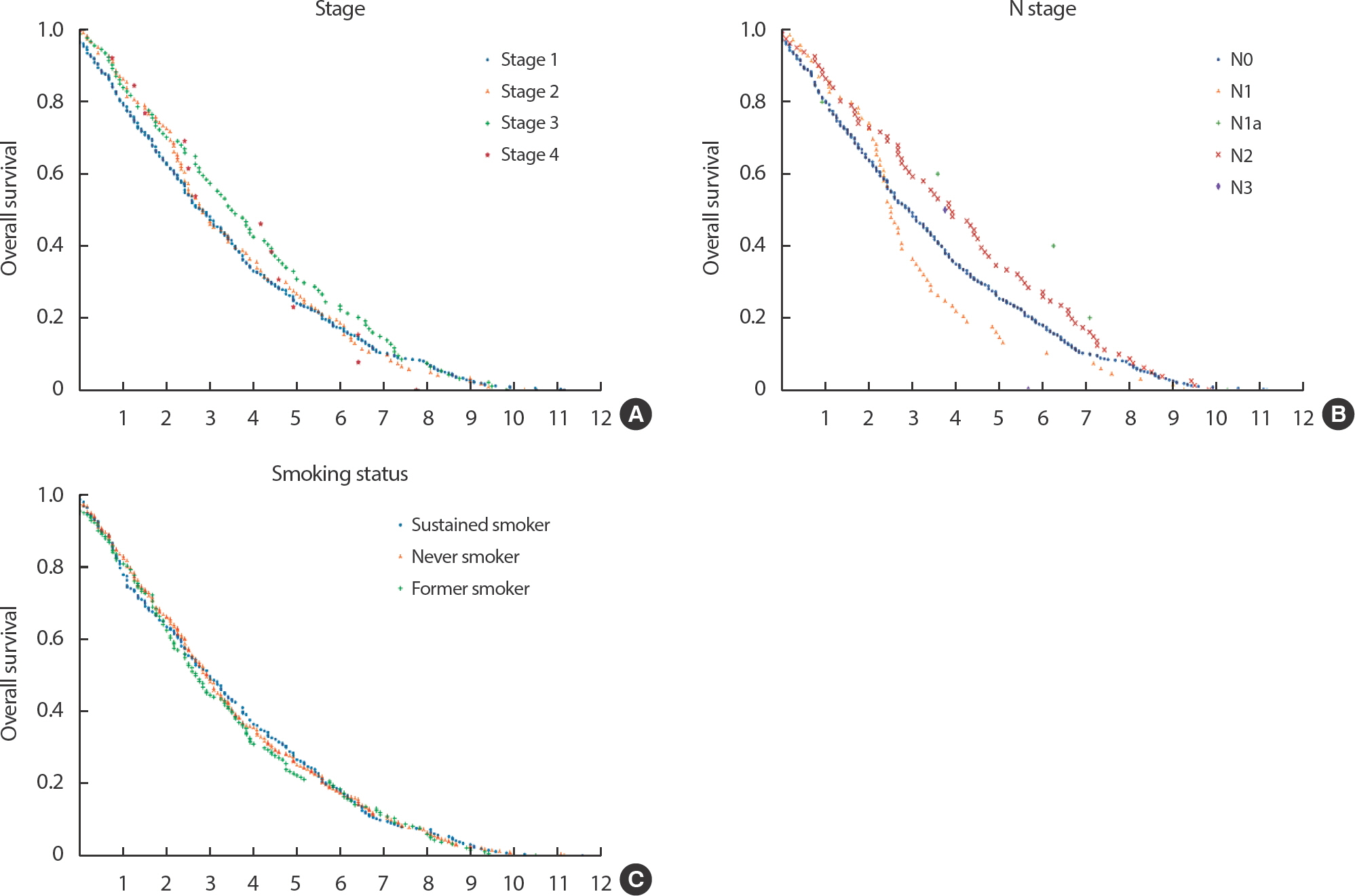
Figure 5.
AbstractObjectivesThis was a pilot study to investigate the possibility of clinical analysis to support the lack of sample size of real data and to generate synthetic data. Since real data has many limitations, such as ethical issues and costly issues, there have been many attempts to create realistic synthetic data. The focus is on whether synthetic data can be used instead of real data.
MethodsThis study analyzed 11,978 lung cancer patients who used anticancer drug therapy using synthetic data as a quasi-experimental study. Clinically significant variables were extracted and some tables containing patient status and treatment records were preprocessed. This experiment was applied to the propensity score matching technique to prevent the bias of covariates. Then, the preprocessed data were analyzed using Kaplan-Meier estimation and Cox proportional hazards model.
ResultsWhen plotting the survival curves, the curves from the synthetic data did not match the curves for the actual data of the other covariates. In Cohort 1, Gen I had a better 5-year OS than Gen II (S1 =0.973, S2 =0.953, p <0.05). Similarly, Gen I anticancer was better than Gen III in Cohort 2 (S1 =0.990, S3 =0.884, p <0.05). In the exploratory subgroup analysis using the Cox regression model, the risk ratio was estimated. We found that Gen I had a better effect on hazard ratio than Gen II and III. However, those results were different from the actual trend.
ConclusionsIt was found that the analysis based on the DATA-FREE-BOX data was different from the trend of the survival analysis conducted with the real data. The trend of this analysis could be different from the real trend. It will be able to contribute to data-validation. Moreover, it is expected that the same methodology can be applied in clinical studies based on actual data by utilizing the technique used in this study.
서 론폐암은 한국에서 시대를 거듭할수록 발병 보고 건수가 꾸준히 증가하고 있고, 모든 암 질환 중에 가장 높은 사망률을 특징으로 하고 있다[1]. 그래서 폐암을 치료하려면 어떤 약물을 처방할지, 예방하기 위해서는 어떤 요인들이 있는지 알아내기 위해 폐암에 대한 다각적인 관점의 연구가 끊이지 않고 있다. 그러나 애석하게도, 폐암 환자의 전자의무기록 데이터(Electronic Medical Record, EMR)는 접근하기 어려울 뿐만 아니라 가공하는 데 시간과 비용이 많이 들며, 데이터가 적재되는 속도에도 명확하게 한계가 있다. 따라서 이러한 단점을 극복하기 위해 실제 데이터인 EMR을 대신하는 실제 데이터를 모방한 합성 데이터(synthetic data)를 이용하여 분석 및 예측모형 학습 등에 활용할 수 있을지 다양한 관점에서의 연구 고찰이 필요하다.
실제로 의료 데이터는 환자의 신상, 질병, 보험 정보 등 다양한 민감 정보를 담고 있어 활용이 쉽지 않다. 데이터 3법이 시행됨에 따라 가명정보를 활용할 수 있는 법적 근거가 마련됐지만, 활용 기관과 사유 등이 명확하지 않고 다양한 규제가 산재해 있어 현실적인 데이터 활용에 어려움을 겪고 있는 실정이다. 이에 적은 양의 실제 데이터를 바탕으로 연구에 활용할 수 있도록 만든 ‘합성 데이터’가 하나의 대안으로 떠오르고 있다. 합성 데이터는 실제로 측정된 데이터를 생성하는 모형이 존재한다고 가정하고, 통계적 방법이나 기계학습 방법 등을 이용해 추정된 모형에서 새롭게 생성한 모의 데이터(simulated data)를 말한다. 개인의 프라이버시를 보호하면서도 민감한 정보를 분석하고자 하는 연구자들에게 데이터를 제공할 수 있는 대안적 개인정보 비식별 조치 기법 중 하나이다. 가령, 원본의 Electronic Health Records (EHR) 데이터셋에서 각 변수들의 공통 분포에 유사한 합성 데이터를 생성하는 AI 모델을 개발해 합성 데이터를 생성하고 재식별 가능성을 최소화하는 시도가 진행되고 있으며[2], 미국 George Mason 대학 연구팀은 오픈소스로 공개된 합성 데이터 생성기 Synthea에서 생성한 메사추세츠 환자 코호트를 대상으로 임상적 중요성이 높은 대장암 검진, 만성 폐쇄성 질환 30일 사망률, 고관절 및 무릎 교체 후 합병증 발생률, 고혈압 관리 등 4가지에 대한 데이터 품질을 측정하여 데이터의 신뢰성이 높은 것으로 확인되었다[3]. 또한, 스위스 제약회사 Roche는 데이터 익명화 전문회사 Statice와 함께 오픈소스로 공개된 Harvard Dataverse 데이터셋을 활용해 임상시험 데이터에서 합성 데이터를 생성하는 연구를 진행하여 합성 데이터가 데이터 분석에 유용하다는 점이 입증되었다.
본 연구에서는 국내 합성데이터를 활용하여 실제 폐암 환자의 항암제 세대별 생존분석에 대한 임상적 연구개발 가능성을 확인하고 이를 통해 합성 데이터의 유용성을 검증하고자 한다. CONNECT의 DATA-FREE-BOX 로부터 암 빅데이터 플랫폼에서 제공하는 암 라이브러리 데이터 활용을 위한 합성 데이터셋을 사용하였다[4]. 구체적인 연구목적은 다음과 같다.
연구 방법연구 개요본 연구에서는 CONNECT DATA-FREE-BOX의 화순전남대학교병원의 데이터를 활용하여 폐암 환자의 합성데이터를 사용하여 변수를 추출 및 변형하여 항암제 세대에 따른 생존 분석을 수행하는 기법과 발전 가능성에 대해 전반적으로 검토하고자 하였다(Figure 1).
연구대상 및 자료수집본 연구에서 사용된 데이터셋은 CONNECT에서 제공하는 DATA FREE BOX 서비스의 합성 데이터이다. 이 데이터셋은 국립암센터와 가천대길병원, 분당서울대학교병원, 연세암병원, 아주대병원, 대구가톨릭대학교의료원, 전북대학교병원, 화순전남대학교병원 총 8개의 기관의 진료기록으로 이루어져 있다. 본 연구는 화순전남대학교병원의 합성 폐암 데이터를 이용하였다(Table 1).
Table 1.Extracting clinical features 해당 테이블 정보에서 필요에 따라 변수의 추출 및 변형을 진행하였다. 환자의 생존기간은 사망일자와 암 진단일자를 기준으로 추정하였으며, 나이의 군집은 65세를 기준으로 계층화하였다. 흡연여부의 경우, 현재와 과거 모두 흡연한 적이 없으면 1 (비흡연자), 과거 흡연한 적이 있으나 현재 흡연하지 않는다면 2 (금연자), 과거 피우지 않았으나 최근 들어 흡연하기 시작한 경우는 3 (흡연 입문자), 과거에도 흡연하였고 현재에도 흡연 중이라면 4 (흡연자)로 계층화하였다. 그리고 암 세포의 종류에 따라 Non-Small Lung Cancer (NSCLC)와 Small Lung Cancer (SCLC)로 분류하였는데, 암 세포의 종류가 ‘ carcinoid’류이면 SCLC로, ‘ carcinoma’류이면 NSCLC로 분류하였다. 최종적으로 학습에 활용된 변수는 ‘ gen’, ‘ survival period’, ‘ event’, ‘ treat purpose’, ‘ sex’, ‘ age’, ‘ job’, ‘ smoking status’, ‘ LCC type’, ‘ LCC pattern’, ‘ LCC differentiation’, ‘ LCC size’, ‘ T descriptor’, ‘ N descriptor’, ‘ M descriptor’, ‘ stage’ 이며, 그 값들은 Table 2에 기술하였다.
Table 2.The meaning of values in variables 항암제 분류분석을 위해서 폐암 치료에 사용되는 항암제들을 작용기전에 따라 1, 2, 3세대로 나누었다. CONNECT에서 제공하고 있는 데이터셋에는 항암제 이름과 코드는 존재하나, 세대 정보는 존재하지 않는다. 따라서, 항암제를 세대별로 분류하기 위해 항암 메커니즘을 조사하였으며 화학적 항암작용/표적 항암작용/면역 항암작용으로 구분하였다. 화학적 항암작용을 하는 항암제를 1세대 항암제로 정의하고, 표적 항암작용을 하는 항암제를 2세대 항암제로, 면역 항암작용을 하는 항암제를 3세대 항암제로 정의하였다(Table 3). 본 연구에서는 1세대 항암제를 이용한 그룹(Gen I)과 2세대 항암제를 이용한 그룹(Gen II), 3세대 항암제를 이용한 그룹(Gen III)으로 표기하였다.
Table 3.Drugs are defined as generation that has their own mechanism 항암제 세대-암 병기별 생존 시간 히스토그램본 데이터셋에 적재된 폐암 환자 중 항암제 세대와 암 병기에 따라 히스토그램을 관찰하였다. 항암제 세대는 Table 2에서 분류한 것을 기준으로 1세대, 2세대, 3세대로 나누어지고, 암 병기는 1기, 2기, 3기, 4기로 나눌 수 있다. 총 12개의 subplot으로 각 경우에 해당하는 사망 환자들의 생존 시간을 구간별로 나누어 몇 명이 분포하는지 관찰하였다.
변수에 따른 생존율 산포도본 연구에서 활용할 수 있었던 변수는 Table 1의 기준으로 선정하였고, 연속적인 값을 가지고 있는 변수와 다변수 범주형 변수도 존재하였다. 이 중 다변수 범주형 변수의 값에 따라 생존율을 계산하고, 생존 시간에 따라 산포도로 나타냈다. 생존율 계산은 Python의 라이브러리인 lifelines에 lifelines.KaplanMeierFitter().survival_function_을 이용하여 계산하였다.
카플란-마이어(Kaplan-Meier) 생존분석생존분석은 대표적으로 생존율을 관찰할 수 있는 Kaplan-Meier analysis와 그 통계적 유의미함을 확인할 수 있는 log-rank test가 있다. 이를 이용하여 특정 집단에 속하는 환자의 5년 생존율을 확인할 수 있다. 그리고 항암제 세대별로 위해비율(hazard ratio, HR)을 관찰하여 두 군 사이의 생존에 대한 우위를 비교하고자 하였다. 이 비교 분석을 통해 어떤 집단이 생존에 효과적이었는지 평가할 수 있으며, 더 나아가 집단이 어떤 특징을 지녔는지에 따라 생존에 좀 더 효과적이었는지 평가할 수 있다.
카플란 마이어 생존 분석은 생존율을 산출하는 방법으로 데이터에서 생존 함수를 추정하는 데 사용되는 비모수 통계 방법이다. 생존기간에 따라 누적생존율을 분석하는 기법으로 누적한계추정법이라고 하기도 한다. 생존기간 t에서 i번째 사건이 발생했을 때 Kaplan-Meier 분석법의 누적생존율은 다음과 같이 정의된다.
di는 사건(- - 1) 번째 사건 이래로 i번째 사건까지 발생한 사망자의 수 로, 이들은 모두 폐암으로 인하여 사망한 경우이다. ni 는 (i - 1) 번째 사건까지 생존한 환자의 수이다. 이 분석법은 생존기간의 분포가 비모 수적이어도 분석할 수 있다는 장점이 있고, 중도절단된 데이터도 고려 하여 분석하기 때문에 이로 인한 편향도 고려할 수 있다.
세 그룹 간의 생존기간이 통계적으로 유의함을 보이기 위해 로그순위 검정(log-rank test)을 통해 유의확률 p를 계산하였다. 귀무가설 H0과 대립가설 H1은 다음과 같이 설정하였다.
이때의 유의수준을 0.05로 설정하여 유의확률 p가 이보다 작을 때 통계적으로 유의하다고 표현한다.
콕스(Cox) 비례위험 모형콕스 비례위험 모형은 생존 시간과 하나 이상의 예측 변수 사이의 연관성을 조사하기 위해 일반적으로 자주 사용되는 회귀 모형이다. 변수의 수가 k 개 존재할 때, 각 변수는 더미 변수 Xi 로 표현(one-hot encoding)되었고, 변수마다 상관계수 bi 가 있다. 이때의 위험함수
이 모형을 사용하기 위해 먼저 각 코호트에서 공변량들에 대해 위험률 이 비례성을 띄는지 살펴보아야 한다[11]. 이것을 비례성 위험 가정이 라고 하는데, 이것을 만족하는지 안하는지 판별하기 위해 log-log survival plot과 Schoenfeld residuals plot 등을 살펴본다. 여기서는 비교군 (Gen II 또는 Gen III)과 대조군(Gen I)의 log-log survival plot만을 살펴 보았다. Log-log survival plot에서는 누적생존율에 대해 log (-log(S))를 취한 뒤 log(t)에 대하여 그 분포를 그려서 두 직선이 평행관계에 놓 여 있으면 비례성 가정이 성립하고 있다고 간주할 수 있다. 본 연구에 서는 Python 라이브러리인 lifelines에서 클래스 KaplanMeierFitter()에 있는 메서드 plot_loglogs()를 통해 두 직선의 평행관계를 확인한다.
그리고 변수들에 대해 전처리가 필요하다. 일부 변수는 범주형 변수여서 바로 더미 변수화하여 사용할 수 있었으나, 연속적인 값을 가지는 일부 변수는 범주형 변수로 변환한 뒤 더미 변수로 만들어 사용하였다. 예를 들어 진단 시 환자의 나이는 65세를 기준으로 하여 그 이상과 미만으로 나누었고, 암 세포크기는 중앙값을 기준으로 하여 그 이상과 미만으로 나누었다.
변수들의 값에 따라 나눈 하위그룹을 다시 1세대와 3세대로 나누어 두 경우를 조사하였다. 1세대와 3세대의 사망자 수와 전체인원 수를 나타냈다. 그리고 1세대 군을 baseline hazard로 설정하여 3세대 군의 위해비율을 산출하였고 95% 신뢰구간(confidence interval, CI)도 함께 나타냈다. 이것을 시각화할 때 x축을 로그 스케일(log-scale)로 표현하였다. HR과 95% CI는 1을 포함하지 않을 때 두 그룹 중 하나가 생존에 유리하다고 판정할 수 있어, 이러한 분석법은 생존에 대한 우위비교를 해석하기 쉽게 해준다. 이때의 우위비교가 통계적으로 유의함을 보이기 위해 유의수준 0.05로 설정하여 유의확률이 이보다 작을 경우 귀무가설을 기각하여 대립가설을 채택한다. 이 때의 귀무가설과 대립가설을 다음과 같이 주어진다.
마지막으로 1세대와 3세대의 누적생존율이 전체 기간 중 총 변화량의 1/2만큼 감소하였을 때의 누적생존율 ΔOS50 과 그때의 생존기간 t를 함 께 표시하였다. ΔOS50 과 t는 Kaplan-Meier survival analysis를 통해 산 출한다. 본래 생존기간을 표현할 때 주로 누적생존율의 중앙값을 이용 하여 이와 함께 사분범위(interquartile range, IQR)를 함께 나타낸다. 그 러나 Figure 3에서 볼 수 있듯이 항암제 세 그룹 모두 관찰기간 중에 누적생존율이 0.5 미만으로 내려가지 않는다. 따라서 누적생존율의 중 앙값을 활용할 수 없었다. 그래서 누적생존율의 총 변화량의 중앙값 (ΔOS50) 과 이때의 생존기간을 나타내는 것으로 대체하였다.
성향 점수 매칭무작위화는 코호트 연구에서 중요하고 필수적인 요소이다. 무작위성이 보장될 때 항암제 세대별에 의한 생존에 미치는 영향을 편향없이 관측할 수 있기 때문이다. 그렇지 않다면 공변량들의 관계에 따라 생존에 미치는 영향을 줄 수 있고, 이로 인해 편향이 발생할 수 있다[12]. 그러나 본 연구는 후향적 연구이기에 무작위성을 보장할 수 없다. 따라서 무작위화에 준하는 효과를 얻기 위해 성향 점수를 이용하여 매칭하는 방법을 사용할 것이다. 성향 점수는 로지스틱 회귀분석을 사용하여 얻을 수 있다. 본 연구에서는 항암제 세대라는 공변량을 one-hot encoding을 통해 이진화(binarization)한 것을 종속변수 Y로, 다른 공변량들을 독립변수 X로 설정해 로지스틱 회귀분석으로 각 세대에 대한 추정 확률을 얻을 수 있다. 이때 독립변수를 설정할 때 몇 가지 유의할 점이 있는데, 공변량 X-Y 간의 독립표본 T-검정을 이용하여 종속성이 있는 경우는 독립변수에서 제외한다. 이렇게 선별된 공변량 간의 추정 확률을 통해 각 비교 군과 대조 군에서 비슷한 추정 확률을 가진 대상끼리 1:N으로 대응시킬 수 있다. 각 군의 추정 확률의 차이는 마치 거리처럼 표현되는데 이 거리를 이용하여 stratified matching, nearest neighbor matching, radius matching 등 여러 matching 방법을 사용하여 대응할 수 있다. 본 연구에서는 N=1로 설정하였고 K-nearest neighbors matching을 이용하여 대응하였다. 이 작업은 Python에서 psmpy라는 라이브러리를 활용하였다[13,14].
본 연구에서는 두 코호트로 나누어 연구를 진행하였고, 각 코호트마다 매칭 과정에서 사용한 변수는 상이하였다. ‘1세대 대 2세대’ 코호트는 Age, Smoking status, LCC type, LCC size, T descriptor, N descriptor, Stage 변수를, ‘1세대 대 3세대’ 코호트는 Age, LCC type, LCC size, N descriptor, M descriptor, Stage를 사용하였다.
연구 결과분석데이터본 연구는 CONNECT DATA-FREE-BOX의 화순전남대학교병원의 폐암 환자 11,978명의 데이터를 대상으로 분석을 실시하였다. 처방을 받은 약물이 폐암 치료를 위해 사용된 것이 아닌 환자 771명을 제외하였으며, ‘ survival period’가 음수로 나온 환자와 같이 비정상 데이터를 분류하여 817명을 제외하여 10,390명의 환자 데이터를 확보하였다(1세대: 8,818명, 2세대: 1,125명, 3세대: 447명) (Figure 2).
사망자분포: 항암제 세대 및 암 병기별 분석폐암으로 인해 사망한 환자의 생존기간을 항암제 세대와 암 병기별로 그 분포를 살펴보았다. 합성 데이터의 이슈로 생존 기간이 음수로 나오는 데이터도 존재하였다. 이번에는 각 병기-세대별 사망자의 수를 관찰하기 위해, 이러한 점을 무시하고 암 병기를 1기부터 4기까지 순서대로 나열하면 1세대가 705건(63.3%)/217건(19.5%)/174건(15.6%)/18건(1.6%)이었고, 2세대가 78건(68.4%)/11건(9.6%)/19건(16.7%)/6건(5.3%)이었으며, 3세대가 98건(71.5%)/16건(11.7%)/19건(13.9%)/4건(2.9%)이었다. 암 병기가 4기에 가깝거나 3세대 항암제로 갈수록 해당 하위그룹의 샘플 수가 적어지는 경향이 있었다. 여기서 암 병기 1-2기인 환자를 전기 암환자로 정의하고 3-4기인 환자를 후기 암환자로 정의하면, 전∙후기 암환자의 수 비율을 비교했을 때 1세대는 82.8% vs 17.2%이고 2세대는 78.0% vs. 22.0%이고 3세대는 83.2% vs. 16.8%이었다. 여기서 3세대 항암제는 실제 임상에서 platinum-based chemotherapy가 실패했을 때 3B 기 환자들에게 처방된다[15]. 즉, 주로 후기 폐암 환자에게 처방되는 경향이 있다. 그런데 본 데이터셋에서는 3세대 항암제 그룹임에도 불구하고 전기(1-2기)인 환자들이 후기인 환자들보다 더 많이 분포하였다. 최근에는 3세대 항암제도 1-3기인 초기 NSCLC 환자에게도 폭넓게 적용하려는 움직임이 있지만, 아직 입증하지 못한 것이 많다. 따라서 초기 NSCLC 환자에게도 면역항암제를 처방하는 것은 흔한 경우는 아닐 것이다. 그래서 본 데이터셋이 현실 데이터셋과 다른 경향성을 가지고 있음을 유추할 수 있었다(Figure 3).
생존분석본 연구에서는 성향 점수 매칭을 적용하여 ‘1세대 대 2세대’ 코호트와 ‘1세대 대 3세대’ 코호트로 구분하였다. ‘1세대 대 2세대’ 코호트는 1세대와 2세대의 비교를 진행하였고 이 때 성향 점수 매칭을 위해 사용된 변수는 Sex, Age, Smoking status, LCC type, LCC size, T descriptor, N descriptor, Stage이다. ‘1세대 대 3세대’ 코호트는 1세대와 3세대와의 비교를 진행하였고 성향 점수 매칭에 사용된 변수는 Sex, Age, LCC type, LCC size, N descriptor, M descriptor, Stage이다.
병기 3기인 환자의 생존율이 1기, 2기, 4기에 비해 천천히 떨어졌다. 병기 4기인 환자는 샘플 수가 다른 병기의 환자보다 적어서 다른 병기 군과의 비교가 어려웠다. 병기는 폐암 환자의 생존율이 감소하는 정도를 보여주는 요인 중 하나이다[16] (Figure 4A).
Figure 4.Survival curve of lung cancer patients at CONNECT (x-axis : survival period [year], y-axis : overall survival). 
N descriptor가 N3에 가까울수록 5년 생존율이 급격하게 감소하려는 경향이 있고, 이는 N descriptor가 환자들의 생존시간에 영향을 끼치고 있음을 알 수 있다[17]. 그러나 본 데이터셋에서는 N descriptor에 따라 생존곡선이 갈라지는 모습을 보였지만, 실제와 같이 높은 N descriptor 환자 군이 낮은 군보다 생존곡선이 빨리 떨어지는 경향을 보이지 않았다(Figure 4B). Smoking status는 현재 및 과거 흡연 여부 정보를 이용해 정의한 것으로, 실제 데이터에서는 현재 흡연 습관을 가지고 있는 사람들은 비흡연자에 비해 폐암으로 인한 사망률이 높다는 것이 보고된 바 있다[18]. 흡연은 폐암으로 인한 사망에 있어서 중요한 요인으로 여겨지는데, 본 데이터셋에서는 그러한 경향을 따르지 않았다(Figure 4C).
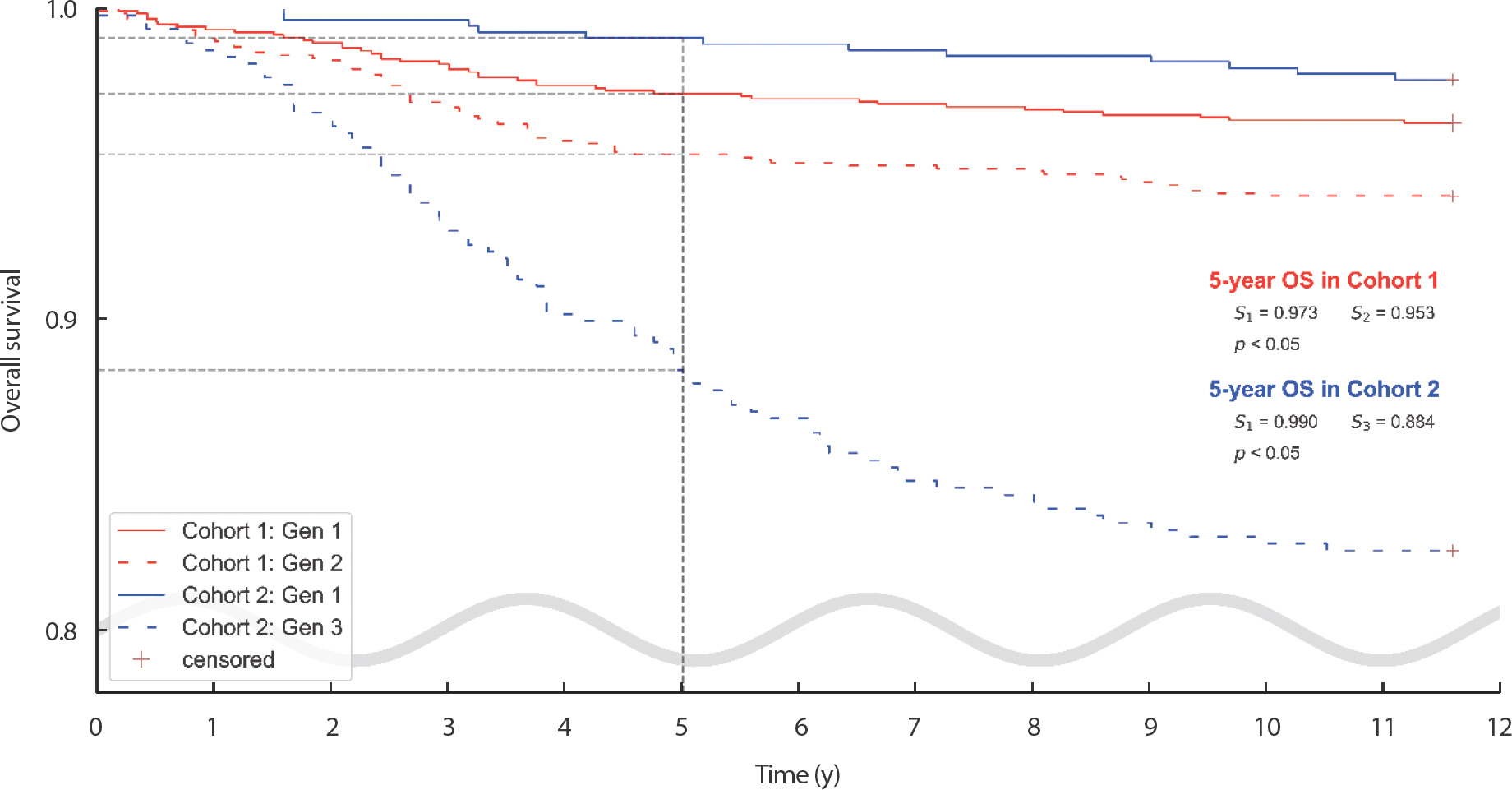
두 코호트 연구에 대하여 각각 Kaplan-Meier estimation을 적용하여 생존분석을 진행하였다. 1세대 대 2세대를 비교했을 때, 5년 생존율이 1세대가 2세대보다 높았다. 이 결과에 대한 차이는 통계적으로 유의미하였다. 그리고 1세대와 3세대를 비교했을 때에도 결과는 비슷하였다. 이 때의 5년 생존율은 1세대가 3세대보다 높았으며, 통계적으로 유의미하였다(Figure 5).
Figure 5.In the Cohort ‘Gen I VS III’, Overall survival (OS) was plotted using Kaplan-Meier analysis. P-value was estimated by log-rank test. 
고 찰본 연구는 폐암 환자의 합성 데이터를 활용하여 생존분석을 진행하였고, 이를 분석한 결과가 실제 임상데이터와 유사한 경향을 가지는지에 초점을 두고 관찰하고자 하였다. 실제 암 환자의 데이터를 활용하기 위해서는 데이터를 오랜 기간 동안 적재 및 보관하고, 이를 열람하려면 환자의 안전을 위해 윤리적인 연구 절차가 선행되어야 한다. 이 절차들은 환자의 정보보호와 안전을 위해 중요하지만, 연구에 있어서 많은 시간을 할애해야 하는 부수적인 절차이다. 그런데 이런 실제 데이터와 비슷한 경향성을 가진 합성 데이터를 활용한다면 예측모형 등을 제작할 때 비용적인 문제와 윤리적인 문제를 최소화하면서 복잡한 절차를 간소화하여 연구에 대한 소요시간과, 금전적 비용을 줄여줄 수 있을 것이다.
합성 데이터의 유효성에 대한 관점으로 보았을 때, 합성 데이터가 실제 데이터의 경향을 참고하고 만들어졌다고 하더라도 본 데이터셋을 통해 볼 수 있는 분석 결과는 현실적인 데이터 바탕의 분석 결과와 많은 차이가 있었다. 예를 들어, N descriptor는 그 값에 따라 폐암 환자의 5년 생존율이 다름을 확인할 수 있었다. 실제로 CONNECT에서 제공되고 있는 합성데이터 분석을 통해 우리는 합성데이터가 아직은 임상현장의 실제 데이터를 대체할 수는 없다는 것을 재차 확인할 수 있었다. 기존 임상 데이터를 기반으로 발표된 암 병기 정보 혹은 흡연 여부에 따른 생존분석 패턴[16,19,20]과 동일한 결과를 도출할 수 없었기 때문이다. 따라서 좀 더 실제 임상 데이터의 대안이 될 수 있는 다양한 방법에 대한 고려가 필요할 것이다. 첫째, 합성 데이터와 실제 데이터에 대한 비교 근거를 정량적으로 설명할 수 있는 적절한 모형의 도입이 필요하다. 예를 들면, Kaplan-Meier curve는 누적생존율의 형태로 표현되기 때문에 exponential function의 형태로 근사할 수 있다. 그러므로 이 점을 이용하여 실제 데이터와 합성 데이터의 차이를 설명할 수 있을 것이다. Cox proportional hazard model을 이용하여 y=λ e−bx의 꼴로 나타낼 수 있는데, 합성 데이터를 만들 때 사용한 실제 데이터를 분석하여 얻은 λ, b, c값과 합성 데이터로 분석한 λ, b, c값의 차이를 비교하면 합성 데이터의 품질을 설명할 수 있을 것이다. 둘째, 데이터 표준화 또한 함께 고려되어야 할 것이다. 본 데이터셋의 TNM 병기 데이터는 American Joint Committee on Cancer (AJCC)의 staging guideline의 버전[16,19,21]에 따라 표기를 변환하는 과정이 수반되었으며 이로 인하여 합성을 통해 얻을 수 있을 정보가 손실될 수 밖에 없었다고 생각된다. 셋째, 모두가 수용 가능한 임상 임계값을 설정하기 위해서는 임상전문의들과의 협의 및 합의 기반 절차가 수반되어야 할 것이다[22]. 마지막으로 사건발생 수나 샘플 수가 어느 정도 확보될 수 있도록 다양한 시도가 필요할 것이다.
또한, 합성 데이터는 그 기초가 되는 원천 데이터의 경향을 모방하여 원천 데이터의 값에 근사하게 제작된다. 따라서 데이터의 변수 간 상관성 등이 원천 데이터의 성질과 비슷하게 나타날 수 있다[3,23]. 그러므로 합성 데이터는 그 원천 데이터가 여러 곳에 유래될수록 일반적인 경향성을 내포할 수 있다. 이런 성향으로 인해 합성 데이터의 모체인 원천 데이터의 변수들의 분포 및 상관관계 등 데이터의 경향성에 대해 임상에서의 상황을 대변할 수 있는지 질적으로 면밀한 검토가 이루어져야 하고, 합성 데이터의 경향이 원천 데이터의 이러한 경향과 얼마나 차이가 나는지 정량적으로 분석할 필요가 있다. 본 연구에서는 사용할 수 있었던 데이터셋이 화순전남대학교병원의 합성 폐암 데이터셋만이 존재하였고, 이것의 원천 데이터는 공개되지 않았기에 본 합성 데이터셋의 질적인 검토에 있어서 한계점이 있었다. 그래서 본 데이터셋의 경향성만으로 임상현장에서의 실제 경향성을 대표한다고 하기에는 무리가 있었다. 이번 연구는 합성 데이터로 임상 실제 데이터의 경향성을 비슷하게 모방하는지 관찰하는 것에 의의가 있다. 후속 연구에서는 데이터셋의 유래를 다양하게 하여 준비할 필요가 있다.
더 나아가 본 연구에서의 한계점을 돌파하기 위해 두 가지 개선사항의 검토가 필요하였다. 첫째, 추가적으로 합성 데이터를 활용한 분석 결과는 통계학적인 관점뿐만 아니라 임상적인 관점에서도 함께 고려하여 결과를 재해석해야 한다. 이를 뒷받침하는 사례로, 통계적 차이뿐만 아니라 임상적 중요도라는 개념을 도입하여 다각적으로 결과를 해석할 수 있음을 보여준 연구들이 존재하였다[24,25]. 이러한 개념을 본 연구에 적용하자면, 95% 신뢰구간의 한계선까지 임상적 의미의 기준 값을 넘어갈 때 비로소 어떤 세대의 항암제보다 좋다고 해석하는 것이 가능해진다. 다만, 임상적 중요도라는 개념은 연구자의 주관성이 반영되는 것이기 때문에 해당 분야에 대한 이해가 많은 임상전문의들과 논의하여 설정하여야 한다. 둘째, 특정 하위그룹에서 샘플의 수가 지나치게 적었다. Cox 회귀 모형은 준모수적인 데이터를 다룰 수 있는 특징이 있지만, 너무 적은 데이터로는 통계적 유의미함과 상관없이 결과를 해석하기 힘들다고 사료되었다. Cox regression analysis에서 보편적으로 변수당 사건발생 수(events per variable, EPV)는 최소 10건으로 설정하지만, 사실 EPV의 최소한계는 데이터를 분석함으로써 알아내야 하고, 일반적으로 20건이 넘을 때 회귀계수의 편향을 제거할 수 있다고 하였다[26]. 따라서 EPV를 고려하여 데이터 수를 확보할 필요가 있다.
결 론본 연구에서는 폐암 환자의 생존분석 연구를 통해 합성 데이터의 활용 가능성을 고찰하고자 하였다. 합성 데이터를 통해 간소한 절차의 임상 데이터를 확보하고 분석하여 연구할 수 있는 기회의 장이 마련되었지만 실제 데이터의 대안으로 그 유용성을 평가하기에는 많은 보완이 필요할 것으로 사료된다. 이와 같은 차이를 지속적으로 연구하여 보완할 수 있는 가이드라인을 개발할 수 있다면 향후 합성 데이터 생성 및 그 데이터의 유효성 검사의 기준을 마련함에 이바지할 수 있을 것이다. 또한, 이러한 연구 시도들이 궁극적으로는 실제 임상 데이터 활용의 파일럿 연구로의 토대 마련에 기여할 수 있을 것이라 기대한다.
REFERENCES1. National Cancer Information Center. Availabe at https://www.cancer.go.kr/ [accessed on August 27, 2022].
2. Differential Privacy: A Q&A With NIST's Mary Theofanos. Available at https://www.nist.gov/blogs/taking-measure/differential-privacy-qa-nists-mary-theofanos [accessed on October 27, 2022].
3. Chen J, Chun D, Patel M, Chiang E, James J. The validity of synthetic clinical data: a validation study of a leading synthetic data generator (synthea) using clinical quality measures. BMC Med Inform Decis Mak 2019;19(1):44. DOI: 10.1186/s12911-019-0793-0.
4. Service ‘ DATA-FREE-BOX’ at CONNECT. Available at https://www.bigdata-cancer.kr/ncc/dataFreeBoxApply.do [accessed on August 30, 2022].
5. Nagano T, Tachihara M, Nishimura Y. Molecular mechanisms and targeted therapies including immunotherapy for non-small cell lung cancer. Curr Cancer Drug Targets 2019;19(8):595-630. DOI: 10.2174/1568009619666181210114559.
6. Gandhi L, Rodriguez-Abreu D, Gadgeel S, Esteban E, Felip E, De An-gelis F, et al. Pembrolizumab plus chemotherapy in metastatic non-small-cell lung cancer. N Engl J Med 2018;378(22):2078-2092. DOI: 10.1056/NEJMoa1801005.
7. Reck M, Rodriguez-Abreu D, Robinson AG, Hui R, Csoszi T, Fulop A, et al. Pembrolizumab versus chemotherapy for PD-L1-positive non-small-cell lung cancer. N Engl J Med 2016;375(19):1823-1833. DOI: 10.1056/NEJMoa1606774.
8. Hellmann MD, Ciuleanu TE, Pluzanski A, Lee JS, Otterson GA, Aud-igier-Valette C, et al. Nivolumab plus ipilimumab in lung cancer with a high tumor mutational burden. N Engl J Med 2018;378(22):2093-2104. DOI: 10.1056/NEJMoa1801946.
9. Gadgeel S, Rodriguez-Abreu D, Speranza G, Esteban E, Felip E, Do-mine M, et al. Updated analysis from KEYNOTE-189: pembrolizumab or placebo plus pemetrexed and platinum for previously untreated metastatic nonsquamous non-small-cell lung cancer. J Clin Oncol 2020;38(14):1505-1517. DOI: 10.1200/JCO.19.03136.
10. Paz-Ares L, Luft A, Vicente D, Tafreshi A, Gümüş M, Mazières J, et al. Pembrolizumab plus chemotherapy for squamous non-small-cell lung cancer. N Engl J Med 2018;379(21):2040-2051.
11. Bae SH. Study on assessing the proportional hazard assumption in the Cox model [dissertation]. Yonsei University, Korea,.2012.
12. Reiffel JA. Propensity-score matching: optimal, adequate, or incomplete. J Atr Fibrillation 2018;11(4):2130. DOI: 10.4022/jafib.2130.
13. Lee DK. An introduction to propensity score matching methods. Anesth Pain Med 2016;11(2):130-148. (Korean).
15. Special cancer immunotherapy. Available at https://www.jkshp.or.kr/journal/view.html?uid=1639&&vmd=Full [accessed on August 27, 2022].
16. American College of Surgeons. Available at https://www.facs.org/me-dia/j30havyf/ajcc_7thed_cancer_staging_manual.pdf [accessed on August 27, 2022].
17. Rami-Porta R, Asamura H, Travis WD, Rusch VW. Lung cancer - ma-jor changes in the American Joint Committee on Cancer eighth edition cancer staging manual. CA Cancer J Clin 2017;67(2):138-155. DOI: 10.3322/caac.21390.
18. Schabath MB, Cote ML. Cancer progress and priorities: lung cancer. Cancer Epidemiol Biomarkers Prev 2019;28(10):1563-1579. DOI: 10. 1158/1055-9965.Epi-19-0221.
19. van Meerbeeck JP, Janssens A. The seventh tumour-node-metastasis staging system for lung cancer: Sequel or prequel? EJC Suppl 2013;11(2):150-158. DOI: 10.1016/j.ejcsup.2013.07.025.
20. Song H, Yoon SH, Kim J, Kim J, Lee KW, Lee W, et al. Application of N descriptors proposed by the international association for the study of lung cancer in clinical staging. Radiology 2021;300(2):450-457. DOI: 10.1148/radiol.2021204461.
21. Detterbeck FC. The eighth edition TNM stage classification for lung cancer: What does it mean on main street? J Thorac Cardiovasc Surg 2018;155(1):356-359. DOI: 10.1016/j.jtcvs.2017.08.138.
22. Page P. Beyond statistical significance: clinical interpretation of reha-bilitation research literature. Int J Sports Phys Ther 2014;9(5):726-736.
23. Das Sarma A, Parameswaran A, Garcia-Molina H, Widom J. Synthe-sizing view definitions from data. In ICDT ‘10: Proceedings of the 13th International Conference on Database Theory;.2010;89-103. DOI: 10.1145/1804669.1804683.
24. Howard AF, Goddard K, Rassekh SR, Samargandi OA, Hasan H. Clini-cal significance in pediatric oncology randomized controlled treatment trials: a systematic review. Trials 2018;19(1):539. DOI: 10.1186/s13063-018-2925-8.
|
|
||||||||||||||||||||||||||||||||||||||