담도암 miRNA 진단 칩을 이용한 분석적 성능 시험
Abstract
Objectives
This study’s purpose was to analyze the analytical performance test of miRNA diagnosis chip for a national project, Development of Multiple Biomarkers Using Integrated Analysis of Next Generation Bio-data, and it was attempted in June 2016.
Methods
The evaluation indices of the analytical performance test were reproducibility, repeatability, and specificity. The analytical performance test of miRNA diagnosis chip for bile duct cancer were subjected to analysis of five markers. One same subject had two sessions of the test, and three times were repeated per each session for reproducibility analysis whereas two different subjects had two sessions of the test, and three times were repeated per each session for repeatability analysis. The measured value taken from six times repeated test of patient group of bile duct cancer and the measured value taken from two times repeated test of normal group were compared by using hierarchical clustering analysis in order for specificity analysis.
Results
As a result of analysis, all CV (coefficient of variance) values of markers were 10% or below for reproducibility and repeatability, and patient group of bile duct cancer were significantly divided from normal group being completely different groups.
Conclusions
Analytical performance test confirmed suitability of miRNA diagnosis chip designed for diagnosing bile duct cancer.
Key words: MicroRNA, Bile duct cancer, Analytical performance, Reproducibility, Repeatability, Specificity
서 론
최근 내시경과 같은 진단 장비의 발전과 보편화로 인하여 조기 암 진단율이 50%에 달하면서 생존율 또한 현저히 증가하고 있다. 그러나 췌장암과 담도암의 경우는 아직까지 조기 진단이 어렵고 예후가 불량하여 여전히 사망률이 높은 것으로 나타나고 있다. 현재까지 췌장암과 담도암의 경우 표준치료 방법으로 알려져 있는 것은 젬시타빈(Gemcitabine)와 같은 항암치료 방법이 유일하다[ 1]. 이와 같은 이유로 근래에는 암의 조기 진단을 높이는 방안으로 바이오마커(bio-marker)와 같은 기술들에 관심이 높아지고 있다[ 2]. 바이오마커와 같은 기술은 사전에 암의 발생 예측이 가능하고 치료반응을 예측할 수 있는 장점이 있으며 개인별 특성에 맞는 치료 및 진단이 가능하다는 것이 가장 큰 장점이다[ 3, 4]. 이전 연구에 의하면 혈액을 이용한 miRNA (microRNA)의 비정상적인 발현은 유방암, 대장암, 폐암, 전립선암 등의 발생과 관련이 있다[ 5]. miRNA는 세포주기, 분화, 발달, 대사 및 노화와 같은 근본적인 생물학적 과정에 관여하고 있다[ 6]. 이와 같은 miRNA를 이용한 진단 칩은 화학물질에 변화되는 유전자 발현 패턴을 마이크로 어레이/어레이 기술(DNA microarray)를 통하여 분석하는 최신 의료기기 진단 기구이다. 최근 DNA를 이용한 어레이 기술 분야에서는 이미 암 진단, 신약 개발, 미생물 연구 및 유전학 등에 보편적인 기술로 상용화가 되었다[ 7- 9]. 그리고 CA19-9와 같은 혈청 단백질을 분석하는 진단 마커의 경우도 상용화가 되어 보편적으로 질병 진단에 사용되고 있다. 하지만 이러한 CA19-9 마커[ 10]의 경우는 민감도와 특이도가 70% 수준으로 낮고, Lewis A, B 유전자[ 11]가 없는 전세계 5%의 인구에 대해서는 CA19-9를 사용할 수 없는 제한점이 있다. DNA와 CA19-9와 같은 상용화 진단 장비들과는 달리 아직까지 miRNA에 대한 연구는 그렇게 많지 않다. 더욱이 아직까지 miRNA을 이용한 특정 암 진단 칩의 적합성을 확인하기 위한 분석적 성능시험에 관한 연구가 없었다. 이에 본 연구에서는 산업통상부 국책과제 “차세대 생명 정보 통합분석”에서 제작된 프로토타입(prototype) 담도암 miRNA 진단 칩의 분석적 성능 시험을 통하여 재현성, 반복성, 특이도를 확인하고 평가기반을 확립하고자 한다. 이 연구의 결과는 아직까지 특정 암에 대한 miRNA 진단 칩의 표준 가이드라인이 없는 상황에서 실증적인 검증 사례를 제시하고 후속 miRNA 분석적 성능시험 연구에 중요한 자료가 될 것이다.
문헌 고찰
담도암 miRNA 진단 칩
담도암 miRNA 진단 칩은 담도암을 비침습적으로 조기 진단하기 위해서 혈액에 존재하는 혈액순환핵산(circulating nucleic acid)의 한 종류인 담도암 miRNA 5종의 존재여부와 발현양상을 DNA 마이크로어레이 방식으로 측정하고 분석하기 위하여 제작된 진단 기구이다.
miRNA 진답 칩의 구성은 miRNA 검출용 DNA Probe가 부착되어 있는 Slide 형태로 구성된다( Figure 1).
(1) Slide: DNA probe가 집적되어 부착되어 있는 유리판 (2) Array: 1개 샘플 실험을 위한 DNA probe의 한 세트 (3) Probe : 특정 miRNA의 존재 유무와 양을 검출하기 위한 40-60개의 뉴클레오타이드(nucleotide)로 구성된 올리고뉴클레오타이드(oligonucleotide)
miRNA 진단 칩의 작동 원리는 전처리 과정을 통해 염색 물질이 부착된 miRNA (target)와 유리 슬라이드(slide) 위에 고정되어 있는 40-60개 DNA 올리고뉴클레오타이드(probe)와 혼성화 반응을 일으켜서 특정 프로브(probe)와 특정 miRNA의 결합을 유도하고 레이저 스캔을 통해 염색물질에서 나오는 파장의 양을 측정하여 특정 miRNA의 존재유무와 양상을 측정을 하게 된다( Figure 2). miRNA 마이크로어레이 프로브의 구성은 다음과 같다.
(1) Hybridization sequence: 타겟 miRNA와 특이적으로 결합하기 위해서 제작된 10-20개 올리고뉴클레오타이드(oligonucleotide) (2) Extended hairpin: Probe-target 간의 결합의 특이성을 높이기 위한 구조 (3) miRNA target: 형광물질이 결합된 miRNA.
분석적 성능시험 지표
분석적 성능시험의 대표적인 평가 지표는 정밀도(재현성, 반복성), 분석적 특이도, 분석적 민감도, 정확도를 들 수 있다.
정밀도는 재현성과 반복성으로 나누어진다. 재현성 시험은 서로 다른 두 명의 실험자에 의한 동일한 시료에 대하여 각각 실시한 실험 결과가 동일한지 검증하였다. 반복성 시험은 동일한 실험자가 동일한 조건에서 3회 반복으로 실험을 실시하여 검증하였다.
분석적 특이도는 교차반응과 간섭반응으로 나누어지며 교차 반응은 담도암이 아닌 정상으로 확진 받은 일반인의 혈액으로 3회 반복 실험을 실시하였다. 간섭반응은 담도암으로 확진 받은 환자의 혈액을 pooling한 후 정상으로 확진 받은 일반인의 혈액을 다시 1:1로 pooling하여 3회 반복 실험하였다.
분석적 특이도 분석은 다중 마커 개발에 사용되는 다변량 자료 분석으로 대표적인 방법인 군집분석(clustering analysis)을 사용하였다. 군집분석은 복잡한 자료의 특성을 파악하여 군집들 간의 관계를 분석하는 통계분석 방법이며 여기서는 계층적 군집분석(hierarchical clustering analysis)을 사용하여 각 군집에 속하는 임의의 두 개체들 사이의 거리를 계산하여 가장 유사성이 큰 군집을 묶어 분류하였다.
정확도는 기대치에 근접하는 정도를 판단하는 척도이고 민감도는 측정할 수 있는 물질의 최소량을 판단하는 척도이다. 분석적 정확도와 민감도는 표준물질이 존재하지 않아 임상시험을 통하여 다수의 질환자들 진단 값을 확인해야 하는 관계로 본 연구의 분석적 성능시험 평가 지표의 범위에서는 제외하였다.
연구 방법
연구 대상자 및 기간
본 연구는 2015년 10월 5일 부터 2016년 4월 12일까지 실시한 산업통상자원부 국책과제 연구자 임상시험(담도암환자 101명, 정상군 63명)에서 연구용 상용칩(Affymetrix, miRNA 4.0)을 이용하여 통계적으로 진단력(area under curve, AUC)이 있는 5종의 마커를 선별하였다. 이후 선별된 담도암 마커 5종(miR-1268a, miR-3162-3p, miR-4270, miR-6729-5p, miR-7107-5p)으로 전용 담도암 miRNA 진단칩에 적용하도록 업체(Agilent Technologies)에 의뢰하여 제작하였고 칩 자체에 대한 분석적 성능 시험에 사용하였다. 분석적 성능 평가를 위한 혈액 검체 시료 수집(담도암 환자 n =1, 정상인 n =1)은 연세대학교 의과대학 신촌 세브란스 병원에서 IRB 승인(No. 4-2012-0528)을 받고 진행하였으며 본 연구는 미국 국립보건원 임상시험등록사이트인 Clinical Trials GOV (NCT02807896)에 등록하였다. 수집된 혈액 검체 시료 분석은 miRNA 진단 칩 분석 장비를 보유하고 Korea Laboratory Accreditation Scheme (KOLAS) 인증을 받은 분석 전문 업체(DNA Link Company)에 의뢰하여 진행하였다. 전체 실험과정은 시험의 품질 보증 및 성적서 발급을 위하여 공인시험 기관인 한국기계전기전자시험연구원(Korea Testing Certification)에서 참관하였다.
시험 기준은 miRNA Microarray System with miRNA Complete Labeling and Hyb Kit 시험방법에 따라 진행하였고 시험 환경은 20.4 ºC, 상대습도 24.5%의 조건을 유지하도록 하였다.
자료수집 방법 및 절차
miRNA 정제는 serum separating tube (SSTs)에 채혈한 담도암 환자 1명 및 정상인 1명의 혈액을 4 ºC 보냉팩에 담아 실험실로 이동 후 혈액 tube를 원심분리기로 3,000 r/min에서 20 분간 원심 분리하여 상층액(serum)을 분리하였다. 이후 제놀루션의 serum miRNA purification kit를 이용하여 serum 내의 total RNA를 다음 순서로 분리하였다( Figure 4A).
(1) 200 μL의 혈액을 kit의 tube에 넣은 후 20초간 vortex로 섞어 주었다. (2) 200 μL의 chloroform을 넣고 10초간 vortex로 섞어 주었다. (3) 원심분리기로 4 ºC, 13,000 r/min에서 10분간 원심분리를 하였다. (4) 상층액 600 μL을 회수하고 새로운 튜브에 넣어 800 μL isopropanol과 섞어 주었다. (5) 원심분리기로 4 ºC, 13,000 r/min에서 5분간 원심분리를 하였다. (6) 침전된 RNA pellet을 제외한 상층액을 제거하였다. (7) 1 mL의 70 %의 에탄올을 넣고 원심분리기로 4 ºC, 13,000 r/min에서 2분간 원심분리를 하였다. (8) 상층액을 제거하고, 원심분리기로 4 ºC, 13,000 r/min에서 20초간 원심분리를 한 후 남은 에탄올을 제거하였다. (9) 침전된 RNA pellet을 RNase가 없는 물 7 μL에 섞어 주었다. (10) 발송 전까지 -80 ºC에서 보관하였다. (11) 시험에 적용하기 전, miRNA의 농도는 극미량 분광광도계(Model: Nano Drop)를 이용하여 측정한 후, 100 ng 정량으로 맞춰 주었다.
정제된 miRNA는 분석적 재현성, 반복성, 특이도를 평가하기 위해서 5개의 튜브에는 ‘1st Dilution Labeling Spike-In’으로 라벨링 하고, 5개의 튜브에는 ‘1st Dilution HybSpike-In’으로 라벨링 하였다. 이후 같은 방법으로 2nd Dilution Labeling Spike-In, 2nd Dilution Hyb Spike-In, 3rd Dilution Labeling Spike-In, 3rd Dilution Hyb Spike-In으로 라벨링 하였다( Figure 4B). 라벨링된 샘플을 Water bath 또는 heat block을 이용하여 100 ºC를 유지하도록 하였다. 그리고 Hyb Spike-In solution이 사용되었을 때 17 μl, Hyb Spike-In solution이 사용되지 않았을 때는 18 μL 의 nuclease-free water를 건조된 샘플에 넣도록 하였다( Table 1). 이후 볼텍스 믹서(Voltex mixer)를 사용하여 골고루 섞어준 뒤, 100 ºC에서 5분간 인큐베이션(incubation) 하고, 즉시 5분 동안 얼음물(얼음:물=1:1)에서 보관한다. 튜브 바닥에 물방울을 모으기 위해 원심분리기(centrifuge)를 사용하여 빠르게 회전시켰다.
Hybridization assembly를 준비하고, SureHyb chamber base 내에 깨끗한 게스켓 슬라이드(gasket slide)를 넣고 Hybridzation sample을 gasket well에 천천히 분주한 뒤, 겹쳐진 슬라이드 위에 SureHyb chamber cover를 놓고 잠금쇠 부품(clamp assembly)를 끼워 넣은 뒤, 챔버(chamber) 위에 잠금쇠를 손으로 단단히 조였다( Figure 4C). 이후 55 ºC에서 20시간 동안 oven rotator rack (Model: Agilent-G2545A)을 이용하여 20 r/min 으로 hybridize한 샘플은 마이크로 어레이 세척(microarray wash)작업을 거친 후 분석 장비 Agilent SureScan Microarray Scanner (Model : SureScan Dx Microarray Bundle)를 이용하여 data를 획득하였다( Figure 4D).
자료 분석
수집된 자료는 통계프로그램 엑셀(Microsoft Excel, 2010)을 이용하여 분석적 성능시험의 지표인 재현성과 반복성을 분석하였고, 특이도는 R Software 3.2.3을 이용하여 계층적 군집분석을 실시하였다.
재현성 시험은 서로 다른 2명의 시험자가 실시한 담도암 miRNA 5개의 마커를 miRNA Microarray System을 이용하여 3회 측정한 후 Log2 값으로 변환하고 평균, 표준편차, coefficient of variance (CV)를 측정하였다.
반복성 시험은 동일 시험자가 실시한 담도암 miRNA 진단 칩의 5개 마커를 miRNA Microarray System을 이용하여 3회 측정한 후 Log2 값으로 변환하고 평균, 표준편차, CV를 계산하였다.
적합성을 확인하기 위한 CV 값은 재현성과 반복성에서 분석된 표준편차 값을 평균값으로 나눈 후 100을 곱하여 계산하였다.
적합성을 판단하기 위한 CV 값 기준은 미국 FDA에서 제시한 생체분석적 개발 방법 및 검증에서 제시한 기준(CV 15%)과 Biochip의 일반적 적용 기준(CV 10%)을 고려하여 CV 10% 이하를 적합성 판단 및 성능기준으로 하였다[ 13- 15]. 분석적 특이도 분석은 정상인의 miRNA를 2회 반복한 결과와 환자군의 miRNA를 6회 반복 시험한 결과를 비교하여 담도암 miRNA 5개를 기준으로 각각 miRNA 계층분석 결과로 확인을 하였다. 계층적 군집분석은 정상인의 miRNA와 환자군의 miRNA 개체들 사이의 거리를 계산(Manhattan 거리와 WARD 알고리즘을 사용)하여 가장 유사성이 큰 군집을 묶어나가는 방법으로 분석하였다.
연구 결과
재현성 분석
담도암 miRNA 진단 칩 재현성 시험 결과는 Table 2와 같다. 총 5종의 마커를 서로 다른 실험자가 1차와 2차로 나누어 각각 3회씩 측정한 후 평균값과 표준편차 값을 분석하였고 이후 CV 값을 분석하였다. 평가 지표의 기준이 되는 CV 분석값은 모두 10% 이하의 결과를 보였다.
반복성 분석
담도암 분석 miRNA 진답칩의 반복성 시험 결과는 Table 3과 같다. 총 5종의 마커를 동일한 실험자가 3회 측정한 평균값과 표준편차 값을 분석하였고 이후 CV 값을 분석하였다. 평가 지표의 기준이 되는 CV 분석값은 모두 10% 이하의 결과를 보였다.
분석적 특이도
담도암 miRNA 진답칩의 특이도 시험 결과는 Figure 5와 같다. 총 5종의 마커를 이용하여 담도암 환자군의 miRNA를 6회 반복 시험한 측정값과 일반 정상인군의 miRNA를 2회 반복 측정치 값으로 계층적 군집분석을 실시한 결과 담도암 환자군과 정상인군의 miRNA는 서로 다른 군집으로 확연히 구분되었다.
논 의
본 연구는 산업통상자원부의 국책과제인 “차세대 생명정보 통합분석을 이용한 다중 바이오마커 개발“ 에서 제작된 담도암 miRNA 진단 칩의 분석적 성능시험을 위하여 평가 지표로 재현성, 반복성, 특이도를 분석하였다.
분석 결과 담도암 miRNA 진단 칩은 재현성과 반복성 실험에서 판정 기준이 되는 CV 값 모두 10% 이하의 결과를 보였다. 그리고 특이도 분석에서는 환자군의 miRNA를 6회 반복 시험한 측정치 값과 정상인의 miRNA를 2회 반복하여 시험한 측정치 값으로 계층적 군집분석을 실시한 결과 담도암 환자군과 정상인군의 miRNA는 서로 다른 군집으로 확연히 구분되었다. 이러한 분석적 성능시험의 결과 제작된 담도암 miRNA 진단 칩은 적합하다는 것을 확인할 수 있었다. 또한 이 결과는 mir-1268a는 담관 폐쇄증 환자가 정상인에 비해 유의하게 낮은 발현량을 보였고 mir-3162-3p는 정상인에 비해 담즙 간경변 환자에게서 유의하게 낮은 발현량을 보였다는 이전 연구와도 일치하는 결과를 보였다[ 16, 17]. 아직까지는 miRNA 진단 장비들이 상용화되지 않은 관계로 분석적 성능 시험에 대한 구체적인 표준 가이드라인 마련되어 있지는 않다. 하지만 본 연구의 분석적 성능시험은 의료기관을 통하여 IRB승인을 받아 혈액을 시료로 진행하였으며 분석 및 실험은 KOLAS 인증을 받은 업체에서 진행하였고 전반적인 시험 과정은 공인시험 기관인 한국기계전기전자시험연구원을 참관하도록 하여 품질 보증을 할 수 있도록 하였다. 그리고 해당 기관들의 의료전문가, 분석 전문가, 인증 전문가, 통계 전문가들의 다양한 의견을 종합하여 합리적인 성능 평가와 시험의 오류를 줄일 수 있었다. 현재 miRNA의 진단 칩에 대한 분석적 성능 시험이 전무한 실정을 감안하면 본 연구는 향후 시험 검사를 위한 기초자료로서 활용 가능성이 있다고 사료된다. 그리고 본 연구의 결과는 현재 급속도로 연구되어 지고 있는 miRNA 진단 칩이 특정 질환 진단 칩으로 상용화가 될 경우 그에 따른 분석적 성능시험에 대한 검증 방안으로 제시될 수 있을 것이다.
하지만 본 연구는 몇 가지 제한점이 있다. 첫째 miRNA의 진단 칩 자체에 대한 분석적 성능시험을 진행한 관계로 담도암 진단 칩의 유효성을 검증하기 위해서는 의료기관의 임상시험을 통한 확인이 추가로 필요하다. 둘째, 분석적 성능시험의 평가 지표 중 정확도와 민감도는 임상시험을 통하여 추가로 검증이 필요하다. 셋째, 담도암에 발현하는 총 5종의 마커만을 진단 칩으로 제작하여 분석적 성능시험을 진행한 관계로 다른 타 암에 적용하기 위해서는 해당 진단 칩 제작과 분석적 성능시험이 추가로 필요하다. 넷째, 혈액 시료의 특성상 의료기관에서 분석 업체로 이동하는 과정에서 시료 변질 등에 대한 품질 관리 방안이 추가적으로 검토되어야 할 것이다.
이러한 제한점에도 불구하고 아직까지 담도암 miRNA 진단 칩을 제작하여 분석적 성능시험을 연구한 사례가 없는 가운데 실증적 시험을 통한 분석 및 방향 제시를 하였다는 것에 큰 의미가 있다고 할 것이다. 더욱이 향후 급속도록 발전하는 miRNA에 대한 후속 연구에서 매우 중요한 자료가 될 것으로 사료된다.
결 론
본 연구는 5종의 miRNA 마커를 이용하여 담도암 miRNA 진단 칩을 제작하고 적합성을 확인하고자 분석적 성능 시험을 실시하였다.
결론적으로 담도암 miRNA 진단 칩의 분석적 성능 시험 지표인 재현성과 반복성은 판단 기준값 CV 10% 이하의 결과와 특이도의 판단 기준인 담도암과 일반인의 계층적 군집분석이 확연히 구분이 되어 적합성을 확인할 수 있었다.
오늘날 의료기기의 발달은 상상할 수 없을 정도로 빠른 진화를 하고 있다. 유전체 진단과 같은 헬스 산업의 무한한 발전 가능성으로 인하여 다양한 의료기기에 대한 개발뿐만 아니라 제품을 신뢰할 수 있는 유효성과 안전성, 성능 분석 및 기타 검증들이 보다 정밀하게 요구가 되고 있다. 이런 점을 고려하면 현재에 상용화되고 있는 기기들뿐만 아니라 선행적으로 연구되고 있는 제품 개발에 대해서도 사전에 신뢰성을 획득할 수 있는 다양한 연구가 이루어 질 수 있도록 해야 할 것이다.
ACKNOWLEDGEMENTS
This study was supported by a research grant (10040174) from the Ministry of Trade, Industry and Energy of South Korea in 2016.
Figure 1.

Figure 2.
The agilent human miRNA microarray probe [ 13]. 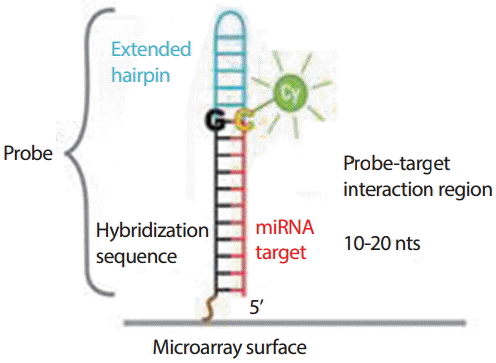
Figure 3.

Figure 4.
Test procedure. (A) Quality control, (B) Labeling, (C) Hybridization, and (D) Scanning 
Figure 5.
miRNA microarray specification of bile duct cancer. 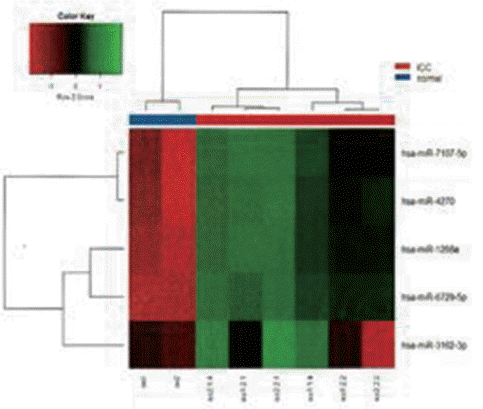
Table 1.
Hybridization mix for miRNA microarrays
|
Components |
|
Volume (μL) |
Total volume (μL) |
|
Used Hyb Spike-In solution |
Labeled miRNA sample |
17 |
45 |
|
3rd Dilution Hyb Spike-In solution |
1 |
|
|
10× Gene Expression Blocking Agent |
4.5 |
|
|
2× Hi-RPM Hybridization Buffer |
22.5 |
|
|
Not used Hyb Spike-In solution |
Labeled miRNA sample |
18 |
45 |
|
10× Gene Expression Blocking Agent |
4.5 |
|
|
2× Hi-RPM Hybridization Buffer |
22.5 |
|
Table 2.
miRNA microarray reproducibility of bile duct cancer
|
miRNA |
1st |
2nd |
Mean |
SD |
CV |
|
1hsa-miR-1268a |
6.69 |
6.81 |
6.75 |
0.09 |
1.30 |
|
2hsa-miR-3162-3p |
3.63 |
3.63 |
3.63 |
0.00 |
0.08 |
|
4hsa-miR-4270 |
9.94 |
10.19 |
10.06 |
0.18 |
1.74 |
|
7hsa-miR-6729-5p |
6.23 |
6.71 |
6.47 |
0.34 |
5.28 |
|
10hsa-miR-7107-5p |
10.26 |
10.42 |
10.34 |
0.11 |
1.10 |
Table 3.
miRNA microarray duplication of bile duct cancer
|
miRNA |
1st |
2nd |
3rd |
Mean1
|
SD |
CV |
|
1hsa-miR-1268a |
6.59 |
7.17 |
6.31 |
6.69 |
0.44 |
6.63 |
|
2hsa-miR-3162-3p |
3.72 |
3.63 |
3.55 |
3.63 |
0.08 |
2.33 |
|
4hsa-miR-4270 |
9.83 |
10.49 |
9.49 |
9.94 |
0.51 |
5.10 |
|
7hsa-miR-6729-5p |
6.23 |
6.75 |
5.70 |
6.23 |
0.53 |
8.47 |
|
10hsa-miR-7107-5p |
10.36 |
10.99 |
9.43 |
10.26 |
0.78 |
7.61 |
REFERENCES
1. Tempero MA, Berlin J, Ducreux M, Haller D, Harper P, Khayat D, et al. Pancreatic cancer treatment and research: an international expert panel discussion. Ann Oncol 2011;22(7):1500-1506.   2. Xu X, Strimpakos AS, Saif MW. Biomarkers and pharmacogenetics in pancreatic cancer. Highlights from the “2011 ASCO Annual Meeting”. Chicago, IL, USA; JOP 2011;12(4):325-329.
3. Huang H, Dong X, Kang MX, Xu B, Chen Y, Zhang B, et al. Novel blood biomarkers of pancreatic cancer-associated diabetes mellitus identified by peripheral blood-based gene expression profiles. Am J Gastroenterol 2010;105(7):1661-1669.   4. Hanas JS, Hocker JR, Cheung JY, Larabee JL, Lerner MR, Lightfoot SA, et al. Biomarker identification in human pancreatic cancer sera. Pancreas 2008;36(1):61-69.   6. Waldman SA, Terzic A. MicroRNA signatures as diagnostic and therapeutic targets. Clin Chem 2008;54(6):943-944.   7. Butte A. 2002. The use and analysis of microarray data. Nat Rev Drug Discov 2002;1(12):951-960.   8. Chung CH, Bernard PS, Perou CM. Molecular portraits and the family tree of cancer. Nat Genet 2002;32(supple):533-540.   9. Stathopoulos A, Levine M. Whole-genome expression profiles identify gene batteries in Drosophila. Dev Cell 2002;3(4):464-465.   10. Koproski H, Steplewski Z, Mitchell K. Colorctal carcinoma antigens detected by hybridoma antibodies. Somatic Cell Genet 1979;5(6):957-972.   11. McCurley RS, Recinos A 3rd, Olsen AS, Gingrich JC, Szczepaniak D, Cameron HS, et al. Physical maps of human alpha (1,3)fucosyltransferase genes FUT3-FUT6 on chromosomes 19p13.3 and 11q21. Genomics 1995;26(1):142-146.   14. Xing WL, Cheng J. DNA Microarray Analysis of Gene Expression Profiles in Hepatocellular Carcinoma, Biochips. Berlin, Germany: Springer-Verlag; 2003. p. 51-59.
15. Manku D, Pak BJ. Flow-through microarrays for rapid biomarker testing. MLO Med Lab Obs 2013;45(4):28-30.
16. Dong R, Shen Z, Zheng C, Chen G, Zheng S. Serum microRNA microarray analysis identifies miR-4429 and miR-4689 are potential diagnostic biomarkers for biliary atresia. Sci Rep 2016;16(6):21084.  17. Tomiyama T, Yang GX, Zhao M, Zhang W, Tanaka H, Wang J, et al. The modulation of co-stimulatory molecules by circulating exosomes in primary biliary cirrhosis. Cell Mol Immunol 2015 September 21;Doi:10.1038/cmi.2015.86 
|
|
















